INTRODUÇÃO
A qualidade da amêndoa tem sido considerada a nível industrial essencialmente com base em parâmetros físicos, como a forma e o peso (Kodad et al., 2006). No entanto, a sua composição química tem igualmente grande importância na qualidade deste fruto seco, altamente nutritivo, rico em ácidos gordos insaturados, proteínas, fibras, compostos fenólicos, vitaminas, como a vitamina E, família de compostos lipossolúveis com forte atividade antioxidante (Barreca et al., 2020). As amêndoas estão entre as melhores fontes de vitamina E dos frutos secos, podendo fornecer cerca de 50% da ingestão diária recomendada com apenas 1 dose de 28 gramas de amêndoa com pele (Institute of Medicine, 2000; Richardson et al., 2009). A sua presença na alimentação humana tem importantes benefícios na promoção da saúde e redução do risco de doenças crónicas, pelo seu papel antioxidante na proteção lipídica contra a oxidação, peroxidação e captação de radicais livres (Galmés et al., 2018). Em termos industriais e de comercialização da amêndoa, a concentração e composição de tocoferol é também um parâmetro igualmente importante para evitar a rancificação durante o seu armazenamento, permitindo aumentar o tempo de prateleira deste produto (Kodad et al., 2018). A vitamina E engloba 4 isómeros (alfa, gama, beta e delta) de tocoferóis e tocotrienois, encontrando-se apenas na amêndoa os α-tocoferol, γ-tocoferol, ß-tocoferol, δ- tocoferol, α-tocotrienol e γ-tocotrienol (Barreira et al., 2012; Çelik et al., 2019). O isómero com maior atividade da vitamina E, o α-tocoferol, é encontrado numa maior concentração na amêndoa, enquanto que os restantes são detetados em pequenas concentrações (Barreira et al., 2012).
A maioria da biossíntese do tocoferol ocorre nos plastídeos com a exceção da síntese do ácido homogentísico (HGA) que ocorre ainda no citoplasma e que serve de precursor à biossíntese dos tocoferóis e trienóis (Fritsche et al., 2017). Os dois substratos HGA e fitil-difosfato (PDP) fundem-se mediados pela enzima vitamina E2/Homogentisate fitil transferase (VTE2/HPT) transformando-se em 2-metil-6-fitil-1,4-benzoquinona (MPBQ) que serve por um lado de substrato à enzima vitamina E1/Tocoferol ciclase (VTE1/TC) para produzir δ-tocoferol, e por outro de substrato à enzima vitamina E3/MPBQ metiltransferase (VTE3/MPBQ MT) para produção de 2,3-dimetil-6-filtil-1,4-benzoquinona (DMPBQ). Este último por sua vez conduz à produção de δ-tocoferol catalisado pela VTE1. Por fim a enzima vitamina E4/γ-tocoferol metiltransferase (VTE4/γ-TMT) catalisa por duas vias a produção do β-tocoferol e do α-tocoferol, a partir do substrato δ-tocoferol ou γ-tocoferol, respetivamente (Georgiadou et al., 2015; Fritsche et al., 2017; Jiang et al., 2017).
Apesar do conhecimento já gerado acerca da cadeia biossintética da vitamina E, a sua regulação molecular ainda é incipiente. Alguns dos genes que codificam as enzimas chaves desta via já foram identificadas em Arabidopsis e em algumas espécies de cereais (Fritsche et al., 2012; Mammadov et al. 2012; Kumar Das et al., 2019), no tomateiro (Quadrana et al. 2013), na oliveira (Georgiadou et al., 2015) e no algodão (Li et al., 2014). No entanto, em amendoeira os genes envolvidos nesta cadeia biossintética ainda não foram caracterizados.
A identificação de variações alélicas associadas a características fenotípicas de interesse tem sido incrementada nos últimos anos pela comunidade científica, com o auxílio de ferramentas moleculares que envolvem técnicas de sequenciação. Os marcadores moleculares têm sido usados na avaliação da diversidade genética, verificação de pedigree, identificação de cultivares, tal como em estudos de associação genética a marcadores de interesse (Mammadov et al., 2012). Por exemplo, os marcadores alélicos associados a genótipos produtores de amêndoa amarga já foram identificados para seleção precoce em amendoeira (Sánchez-Pérez et al., 2010; Ricciardi et al., 2018).
No presente estudo reportamos primeiramente, a identificação e a caracterização de três genes candidatos (PdVTE 2, 3 e 4) envolvidos na cadeia biossintética do tocoferol na amendoreira, seguida da identificação de variações naturais nas regiões do promotor, da Open reading frame (ORF) e da 3’untranslated region (3’-UTR) da sequência PdVTE4, pela primeira vez nas cultivares Antoñeta, Lauranne e Soleta.
MATERIAL E MÉTODOS
Material vegetal
As amostras de folhas de três árvores das cultivares (Antoñeta, Lauranne e Soleta) foram colhidas em outubro de 2019, em plantações localizadas em Ferreira do Alentejo, Beja, Portugal. O material vegetal depois de preparado e macerado em azoto líquido, foi armazenado a -80ºC para as subsequentes análises moleculares.
Extração de DNA
O material vegetal macerado foi usado para extração do DNA genómico, a partir do kit DNAeasy Plant Mini Kit (Qiagen, Alemanha), de acordo com as instruções do fabricante. A qualidade do DNA genómico foi averiguada por eletroforese em gel de agarose (1%), com GreenSafe Premium (NZYTech, Portugal) em Sistema GelDoc XR (BioRad, USA) e por espectrofotometria (Nanovue, Biochrom, USA), através das razões A260/A230. A concentração do DNA genómico foi obtida por espectrofotometria (Nanovue, Biochrom) e fluorometria (Fluorometer Qubit 4, ThermoFisher Scientific, USA).
Desenho de oligonucleótidos e análises in silico
As sequências genómicas do VTE2, VTE3 e VTE4 e respetivas sequências proteicas do Prunus dulcis var. Texas foram identificadas e caracterizadas, a partir do genoma de referência P. dulcis var. Texas (Jung et al., 2019; v.2.0) depositada na base de dados Genome Database for Rosaceae (GDR; https://www.rosaceae.org) e com recurso a ferramentas de BLAST (Basic Local Alignment Search Tool), Expasy Translate tool e BioEdit. A caracterização das sequências genómicas a nível das regiões do promotor, codificante e 3’-UTR foi efetuada para os genes putativos da variedade Texas, tendo em conta a localização cromossómica, o tamanho total da sequência genómica e da região codificante (CDS), o número e tamanho dos exões/intrões e tamanho 3’-UTR.
Para a identificação das novas sequências genómicas do PdVTE4 nas cultivares Antoñeta, Lauranne e Soleta, por PCR e sequenciação, novos oligonucleótidos foram desenhados com recurso ao software Primer3plus (Untergasser et al., 2012), obedecendo a parâmetros específicos, tais como: tamanho dos fragmentos entre 700 e 850 pb, tamanho máximo dos oligonucleótidos de 23 pb, e percentagem de GC com máximo de 50%. O self anneling e a formação de estruturas secundárias foram validados com recurso ao software PCR Primer Stats (Stothard, 2000). A sequência dos oligonucleótidos, o tamanho do fragmento esperado e a temperatura de annealing encontram-se descritos no Quadro 1. O oligonucleótido P3 amplificou o início do promotor. Os respetivos oligonucleótidos foram desenhados de forma a não coincidir com as regiões menos conservadas descritas nas bases de dados, e de maneira a que o oligonucleótido reverse começasse a amplificar após o oligonucleótido forward do fragmento seguinte, criando regiões comuns entre as extremidades dos fragmentos amplificados. No total, seis pares de oligonucleótidos diferentes foram desenhados para cobrir cada sequência genómica total.
Quadro 1 Sequência dos oligonucleótidos e respectivos tamanho do fragmento esperado e temperatura de annealing, para o gene candidato PdVTE4. F: forward; R: reverse; pb: pares de bases
| ID | F/R | Sequência dos olinucleótidos | Tamanho fragmento (pb) | Temperatura de annealing (ºC) |
| VTE4.P1 | F | 5’ - GGAGTGTAATTGGGAACC - 3’ | 786 | 52 |
| R | 5’ - GCGGATCCAGAAATTATTC - 3’ | |||
| VTE4.P2 | F | 5’- CGTCATCTTCCACCTCAAG - 3’ | 731 | 60 |
| R | 5’ - TTGTAGCGTGCAGTTTGAC - 3’ | |||
| VTE4.P3 | F | 5’ - TTTGGCCTCGTCTAACATGA - 3’ | 805 | 60 |
| R | 5’ - CCATAAGCCAGACGACTCG - 3’ | |||
| VTE4.P4 | F | 5’ - TGGGTTGAGGAGATTGAACG - 3’ | 778 | 54 |
| R | 5’ - GCTTTGTCAGGCATGTGTTC -3’ | |||
| VTE4.P5 | F | 5’ - GCTCTGGATCAACCATTTCC - 3’ | 748 | 54 |
| R | 5’ - GTGGGCAAATTGAACAGACA - 3’ | |||
| VTE4.P6 | F | 5’ - CCTTTAGGTTCTGTTTGAAC - 3’ | 755 | 54 |
| R | 5’ - CTTGAGCCGTTGAGGAAC - 3’ |
Amplificação de fragmentos por PCR e sequenciação
O gene PdVTE4 foi amplificado por PCR nas variedades de amendoeiras em estudo através de reações de PCR, com o conjunto de oligonucleótidos descritos no Quadro 1. A amplificação dos fragmentos de DNA por PCR foi realizada usando 0,1-0,5 ng de DNA genómico, 0,5 μM de oligonucleótidos forward and reverse (Quadro 1) e 1x Dream Taq PCR Mastermix (Thermo Scientific, USA), num volume final de 20µl. As reações de PCR foram realizadas no termociclador Life ECO termal cycler (Bioer, China) de acordo com as seguintes condições: 95ºC durante 3 min; 35 ciclos de 30s a 94ºC; temperatura de annealing descritos na tabela 1 durante 30s e 2min a 72ºC, finalizando 10 min a 72ºC. Controlos negativos, sem DNA genómico, foram incluídos em cada reação de PCR.
Os produtos de PCR amplificados foram separados por eletroforese horizontal num gel de agarose 2% com 1µl de GreenSafe Premium (NZYtech, Portugal). Os tamanhos dos fragmentos amplificados foram estimados através do NZYDNA Ladder V (NZYtech, Portugal).
As amostras que apresentaram amplificação de DNA específica de PCR foram selecionadas para sequenciação pelo método de Sanger, na modalidade de outsourcing. As sequências de DNA e de aminoácidos obtidas foram analisados com recurso dos programas BioEdit, Multalin, Expasy e Translate tool.
As sequências genómicas obtidas para os genes PdVTE4 nas variedades Antoneta e Soleta e Lauranne estão disponíveis no NCBI Sequence Read Archive sob os números de acesso: OQ436579, OQ436580, OQ436581, OQ436582, OQ436583, OQ979144 e OQ979145.
RESULTADOS E DISCUSSÃO
As variações genéticas detectadas na sequência genómica podem resultar em alterações fenotípicas num organismo, constituindo potenciais marcadores moleculares. Além disso, as novas tecnologias de sequenciação e a redução significativa do seu custo permitiram um enorme avanço na identificação dos novos marcadores genéticos do tipo SNP e InDel (polimorfismo de inserção/deleção), apresentando-se atualmente como ferramentas importantes em programas de melhoramento. Os SNPs são marcadores bialélicos abundantes nos genomas cuja identificação é passível de automatização, tornaram-se ferramentas importantes nestes programas (Li et al., 2014). Os InDels, resultantes de deleções ou inserções entre 1 a 10 000 pb no DNA genómico, são marcadores altamente polimórficos e adequados para genotipagem baseada em sistemas de electroforese em gel de agarose de baixo custo (Kizil et al., 2020).
Assim, a identificação e caracterização de sequências genómicas específicas de interesse são fundamentais para a deteção destes mesmos polimorfismos. Neste estudo foi possível numa primeira fase, identificar e caracterizar a sequência genómica dos genes putativos PdVTE 2, 3 e 4, e posteriormente identificar no gene PdVTE4, polimorfismos genéticos do tipo SNP e InDels associados às regiões do promotor, da ORF e 3’-UTR presentes nas três cultivares em estudo.
Análises in silico
A análise in silico possibilitou a identificação dos genes putativos PdVTE2, PdVTE3 e PdVTE4, envolvidas na via biossintética do tocoferol na amendoeira. A caracterização dos genes candidatos em P. dulcis var. Texas foi efetuada tendo em conta a localização cromossómica, o tamanho total da sequência genómica e da região codificante (CDS), o número e tamanho dos exões/intrões, tamanho 3’UTR e das respetivas proteínas (Quadro 2). Os genes putativos PdVTE2, 3 e 4 localizam-se nos cromossomas 1, 8 e 7 respetivamente. Possuem uma região codificante que varia entre os 1221 e 1029 pb. O PdVTE2 possui o maior número de intrões (12) e exões (13) comparativamente com as outras duas sequências analisadas. A sequência proteica correspondente é igualmente maior [406 aminoácidos (aa)] na PdVTE2, em relação às sequências do PdVTE3 e 4 (342/3 aa).
Quadro 2 Caracterização da sequência genómica de PdVTE2, PdVTE3 e PdVTE4 e das respectivas proteínas em Prunus dulcis var. Texas. Pb: pares de bases; aa: aminoácidos
| Prunus dulcis var. Texas | |||
| Gene | PdVTE2 | PdVTE3 | PdVTE4 |
| Nome | Vitamina E2 (VTE2) ou Homogentisate fitil transferase (HPT) | Vitamina E3 (VTE3) ou 2-metil-6-fitil-1,4-benzoquinol metiltransferase (MPBQ MT) | Vitamina E4 (VTE4) ou γ-tocoferol metiltransferase (γ-TMT) |
| ID gene | Prudul26A007290 | Prudul26A025531 | Prudul26A011953 |
| Localização cromossómica | Pd01, 6290533 - 6297934 (- strand) | Pd08, 18595950 - 18599125 (+ strand) | Pd07, 13096900 - 13099513 (+ strand) |
| Tamanho total (pb) | 7402 | 3176 | 2614 |
| Tamanho CDS (pb) | 1221 | 1029 | 1029 |
| Promotor (pb) | 313 | 351 | 509 |
| Nº de exões | 13 | 3 | 6 |
| Tamanho Exões (pb) | 43; 45; 287; 84; 75; 84; 90; 102; 96; 72; 96; 105; 42 | 566; 307; 156 | 340; 173; 105; 204; 103; 104 |
| Tamanho Intrões (pb) | 463; 115; 199; 88; 355; 1066; 809; 805; 189; 178; 521; 591 | 430; 272 | 111; 156; 94; 141; 275 |
| Tamanho 3’ - UTR (pb) | 444 | 1094 | 299 |
| ID proteína | Prudul26A007290 | Prudul26A025531 | Prudul26A011953 |
| Tamanho proteína (aa) | 406 | 342 | 343 |
| Função | Fitilação | Metilação | Metilação |
Identificação e caracterização genómica do PdVTE4 por sequenciação
A identificação e caracterização genómica do gene PdVTE4 foi realizada através da sequenciação de fragmentos específicos de DNA, nas cultivares Antoñeta, Lauranne e Soleta. Esta caracterização foi igualmente acompanhada pela identificação de variações naturais na sequência genómica, na população amostrada. No total, foi possível a identificação de sequências de DNA genómico de elevada qualidade a partir de 108 fragmentos, com um tamanho que variou entre 278 e 907 pb por fragmento, para cobrir a sequência do gene PdVET4, nos diferentes genótipos. As novas sequências obtidas por sequenciação de Sanger foram alinhadas contra o genoma de referência de Prunus dulcis var. Texas. A localização cromossómica do PdVTE4 foi identificada no cromossoma 7 (P07: 13095073 a 13095623 +) e as sequências consenso apresentaram uma percentagem de identidade superior a 95% (Quadro 2).
No seguimento desta análise das novas sequências foi possível efectuar uma primeira caracterização das sequências genómicas em cada genótipo da região do promotor, da ORF, com a identificação dos exões e intrões, e da 3’-UTR do gene PdVTE4, em 7 dos 9 indivíduos testados nas cultivares Antoñeta, Lauranne e Soleta (Quadro 3). A sequência genómica total identificada foi de cerca de 2615 pb, com 508 pb para a região do promotor, 1029 a 1052 pb para a região codificante e com 5 diferentes intrões. Apenas num dos indivíduos da Lauranne e da Soleta não foi possível sequenciar a região completa da sequência codificante (CDS), e subsequentemente a completa sequência de aminoácidos.
Quadro 3 Caracterização da sequência genómica do gene PdVTE4, com a identificação do tamanho da região do promotor, da ORF e da 3’UTR e total em pares de base (pb), e da respectiva sequência proteica em aminoácidos (aa), obtido nos 7 indivíduos (ID) avaliados nas cultivares Antoñeta (G2 e G3), Lauranne (G1, G2 e G3) e Soleta (G2 e G3) estudadas neste trabalho e a sua comparação com a cultivar Texas utilizada como genoma de referência
| Cultivar | ID | Total | CDS | Promotor | Exões | Intrões | 3’ - UTR | Proteína | ||||||||||||||||
| Texas (Referência) | 2614 | 1029 | 509 | 340, 173, 105, 204, 103, 104 | 111, 156, 94, 141, 275 | 299 | 342 | |||||||||||||||||
| Antoñeta | G2 | 2615 | 1031 | 508 | 340, 173, 105, 204, 103, 104 | 113, 156, 94, 141, 275 | 299 | 342 | G3 | 2615 | 1051 | 508 | 340, 173, 105, 204, 103, 104 | 113, 156, 94, 141, 275 | 299 | 342 | ||||||||
| Soleta | G1 | NA | NA | 508 | NA, 173, 105, 204, 103, 104 | NA, 156, 94, 141, 275 | 299 | NA | G2 | 2614 | 1031 | 508 | 340, 173, 105, 204, 103, 104 | 111, 156, 94, 141, 276 | 300 | 342 | G3 | 2614 | 1030 | 508 | 340, 173, 105, 204, 103, 104 | 111, 156, 94, 141, 276 | 299 | 342 |
| Lauranne | G2 | 2615 | NA | 508 | 340, 173, NA, 204, 103, 104 | 113, 156, NA, 141, 275 | 299 | NA | G3 | 2615 | 1030 | 508 | 340,173, 105, 204, 103, 104 | 113, 156, 94, 141, 275 | 299 | 342 |
Nota: NA - não determinado
Identificação de polimorfismos no PdVTE4
Sendo a VTE4 uma das enzimas chave na via de biossíntese da vitamina E, transformando o γ-tocoferol em α-tocoferol, este estudo foi posteriormente direcionado para a análise do gene PdVTE4 responsável pela sua codificação. Assim a caracterização das sequências genómicas do PdVTE4 na população em análise de Prunus dulcis permitiu identificar vários polimorfismos putativos dentro dos quais 90,3% do tipo SNP (Single-nucleotide polymorphism) versus 9,7% InDel (Inserção/Deleção).
A partir da população analisada, foram obtidos um total de 28 polimorfismos diferentes do tipo SNP ao longo da sequência de 2615 pb identificada (Figura 1), com uma frequência média de um SNP em cada 93 pb, um em cada 73 pb no promotor e um em cada 257 pb na região codificante. A região intrónica foi a que apresentou um maior número de SNPs nas 3 cultivares estudadas e em contrapartida a região exónica apenas apresentou dois polimorfismos distribuídos em dois indivíduos da cultivar Lauranne e Soleta (Figura 2). A distribuição de SNPs variou também entre os indivíduos amostrados, apresentando-se a cultivar Soleta com uma maior taxa média de ocorrência, um SNP em cada 138 pb, enquanto na Lauranne observou-se uma menor taxa de frequência média, de um SNP entre 227 pb.
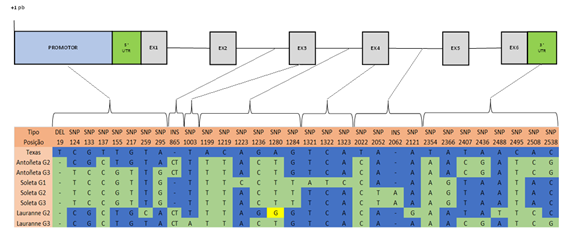
Figura 1 Polimorfismos do tipo SNP e InDel identificados nas regiões do promotor, exões, intrões e 3'UTR, em comparação com o genoma de referência da cultivar Texas. A posição específica de cada polimorfismo está indicado ao longo da sequência genómica do gene PdVTE4, nos sete genótipos analisados de amendoeira, nas cultivares Antoñeta, Lauranne e Soleta.
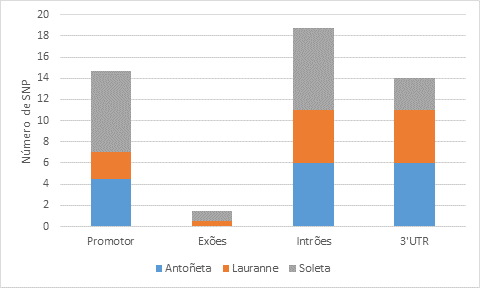
Figura 2: Número médio de SNP identificados nas regiões do promotor, exões, intrões e 3'UTR no gene PdVTE4 nas cultivares Antoñeta, Lauranne e Soleta.
As transições C/T foram a causa mais comum dos SNPs identificados, com uma percentagem de 28,6, enquanto as transversões A/C, G/C e G/T apresentaram uma menor percentagem de 10,7 cada (Figura 3).
No entanto, os SNPs encontrados nas regiões codificantes na Lauranne e Soleta não alteraram a sequência de aminoácidos, tratando-se assim de polimorfismos sinónimos.
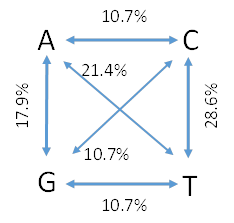
Figura 3: Distribuição de SNPs do tipo transições e transversões identificados na população de amendoeira estudada.
Além dos polimorfismos do tipo SNP, três variações naturais observadas do tipo InDels foram identificadas, tanto a nível de deleções (InDel 1) como de novas inserções (InDel 2 e 3), na região do promotor e nas regiões intrónicas, respetivamente (Figura 1).
Polimorfismos do tipo SNPs e InDels no gene VTE4 já foram associados à diversidade do conteúdo de α-tocopherol e ao total da vitamina E no arroz (Wang et al., 2015), soja (Lipka et al., 2013) e milho (Li et al., 2012; Kumar Das et al., 2019). Neste último, um alelo mutante do ZmVTE4 com uma deleção de 7 e 118 pb na região 5’UTR e promotor, respetivamente, melhorou significativamente o conteúdo de α-tocoferol no grão de milho (Kumar Das et al., 2019). Em Oryza sativa, as variações de nucleotídeos do tipo SNP na região codificante do OsVTE4 foram significativamente associadas às variações do conteúdo de α-tocoferol, enquanto os polimorfismos de nucleotídeos na região promotora do OsVTE4 também puderam demonstrar parcialmente a correlação com as variações no conteúdo de α-tocoferol (Wang et al., 2015).
Outros genes da cadeia biossintética do tocoferol foram igualmente descritos e correlacionados com a diversidade do conteúdo do tocoferol. Cerca de 26 polimorfismos no gene BnaX.VTE3.a foram significativamente associados com características de tocoferol na colza (Fritsche et al., 2012). Enquanto que na cevada alelos do gene VTE2 localizado no cromossoma 7 diferiram na sua capacidade de acionar a produção de vitamina E (Schuy et al., 2019), o que reforça a importância da caracterização destes genes analisados na amendoeira no presente estudo.
Em estudos prévios, a nossa equipa de investigação otimizou a quantificação do teor de tocoferol por HPLC em amostras de amêndoas, e identificou com sucesso o conteúdo de α- e γ-tocoferol em variedades tradicionais e comerciais, obtendo diferenças significativas entre algumas das variedades analisadas (Almeida et al., 2023). Futuramente pretende-se relacionar os polimorfismos já detetados, do tipo SNP e InDel, nos diferentes genótipos e cultivares de amendoeira estudados, e correlacioná-los com o perfil de tocoferol dos mesmos.
Marcadores co-dominantes específicos poderão então ser desenvolvidos a partir dos polimorfismos SNP e InDel para aplicação em programas de melhoramento da amendoeira. Compreender a base genética dessas características será valioso para melhorar a qualidade nutricional ao nível da síntese de vitamina E deste fruto.
CONCLUSÕES
Este trabalho permitiu identificar e caracterizar as sequência genómicas dos genes putativos PdVTE 2, 3 e 4 em P. dulcis. Na população em estudo com as variedades Lauranne, Antoñeta e Soleta, foram detectados cerca de 28 polimorfismos genéticos do tipo SNP com uma frequência média de um SNP em cada 93 pb do gene PdVTE4, e três do tipo InDel resultantes de deleções ou inserções. A identificação dos genes, tal como o PdVTE4, envolvidos na cadeia biossintética do tocoferol e a detecção de suas variações alélicas, na amendoeira, poderão ajudar na compreensão dos mecanismos genéticos que regulam a produção de tocoferóis, constituindo uma base de dados de frequências alélicas, específica para cada uma das populações estudadas, que possa ser aplicado futuramente em programas de melhoramento, na seleção de genótipos mais tolerantes ao stress oxidativo e na produção de amêndoas de qualidade diferenciadora em vitamina E.