Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Arquivos de Medicina
On-line version ISSN 2183-2447
Arq Med vol.23 no.4 Porto Aug. 2009
Hanseníase no Mundo Moderno
O Que Sabemos Sobre a Influência Genética do Hospedeiro no seu Controle?
Danilo Santana Alessio Francheschi*, Willian Sergio do Sacramento*, Priscila Saamara Mazini*, Jeane Eliete Laguila Visentainer†
*Universidade Estadual de Maringá, Brasil; †Departamento de Ciências Básicas da Saúde, Universidade Estadual de Maringá, Brasil
A hanseníase é uma doença infecciosa crônica causada pelo Mycobacterium leprae. Enquanto muitos indivíduos contaminados com o bacilo não apresentam sintomas, outros manifestam formas clínicas distintas. No início, a forma Indeterminada pode se manifestar e evoluir para as forma polares: Hanseníase Virchowiana (HV) ou Hanseníase Tuberculoide (HT). HV representa o pólo onde a resposta imune celular é deficiente e a carga bacilar bastante grande, enquanto HT se caracteriza pela melhor resposta imune celular e controle da carga bacilar. Uma forma com características intermediárias entre os dois pólos também pode ocorrer: Dimorfa ou Borderline. Existem muitos fatores que podem influenciar o desenvolvimento da doença e sua evolução, entre eles, a genética do hospedeiro. O objetivo desta revisão é apresentar os mais recentes estudos sobre a influência da genética do hospedeiro na imunopatogênese da hanseníase.
Palavras-chave: hanseníase; Mycobacterium leprae; genética do hospedeiro.
Leprosy on the Modern World
Leprosy is a chronic infectious disease caused by the Mycobacterium leprae. While many infected individuals with the bacillus do not present symptoms, others manifest different clinical forms of the disease. In beginning, the undetermined form may manifest and evolutes to pole forms: Lepromatous Leprosy (LL) or Tuberculoid Leprosy (TL). LL represents the pole where the cellular immune response is deficient and the bacillary load is high, while TL is characterized by the best cellular immune response to and control of the bacillary load. One form with intermediate characteristics between the two poles may also occur: Borderline. There are many factors that influence the development of the disease and its evolution, among than, genetic factors of the host. The main of this revision is to present the most recent studies on host-genetic influences on leprosy immunopathogenesis.
Key-words: leprosy; Mycobacterium leprae; host’s genetic.
INTRODUÇÃO
Hanseníase é uma doença infecciosa crônica causada pelo Mycobacteriumleprae que primeiramente afeta a pele e os nervos periféricos. Embora a prevalência global da hanseníase tenha diminuído ao longo dos anos, devido à terapia com múltiplas drogas, a detecção de novos casos permanece estável, aproximadamente, 500.000 novos casos todo ano. Atualmente, 80% dos casos novos concentram-se em países localizados na faixa intertropical: Índia; Brasil; Myamar; Madagascar; Nepal e Moçambique (1). Atualmente, o Brasil ocupa o segundo lugar em números absolutos de casos de hanseníase no mundo (2). É um processo infeccioso crônico que, apesar de sua infectividade, é passível de cura, mas depende do grau de endemicidade do meio.
Esta revisão focaliza a sobrevivência do parasita no hospedeiro, a qual está relacionada principalmente a fatores genéticos do próprio hospedeiro, no caso do M. leprae. Diferente de outros microrganismos, ele não possui variabilidade genética suficiente para explicar as distintas sintomatologias que se apresentam. Enquanto muitos indivíduos contaminados com o bacilo não apresentam sintomas, outros manifestam formas clínicas distintas.
O M. leprae foi descrito em 1873 pelo norueguês Am-auer Hansen. É bacilo álcool-ácido resistente e parasita intracelular com predileção pela célula de Schwann e pele (3). O sequenciamento completo do seu genoma revelou extensa redução evolutiva, com perda de grande número de genes em comparação com o M. tuberculosis, desde que essas espécies divergiram de um ancestral comum (4).
A doença apresenta um espectro de sintomas que se manifestam como formas clínicas distintas, as quais têm como principal característica o tipo de resposta imunitária que o hospedeiro apresenta frente ao microrganismo. Duas formas são totalmente opostas, a Hanseníase Tuberculoide (HT) e a Virchowiana (HV) (5). Enquanto, na forma HT o indivíduo manifesta uma resposta imune celular bem desenvolvida, a qual consegue deter a multiplicação do bacilo; na forma HV, a resposta celular é deficitária, levando à multiplicação acelerada do bacilo que continua a ser transmitido se o tratamento não for iniciado (6). Uma forma intermediária a estes dois pólos também já foi caracterizada, na qual os indivíduos apresentam respostas imunes que variam entre os extremos. Esta é denominada de Hanseníase Dimorfa (HD) ou Borderline (5), a qual pode ser classificada como Borderline-Tuberculoide (BT), Borderline-Virchowiana (BV) ou Borderline-Borderline (BB), dependendo do padrão de manifestações clínicas eimunológicas. A forma Indeterminada, geralmente, aparece no início podendo evoluir para a cura ou para qualquer uma das formas já citadas.
Para determinar o tipo de tratamento mais adequado para o indivíduo portador do bacilo e a conduta com relação aos membros da sua família, as formas HV, BV e BB podem ser reclassificadas como multibacilares e as HT e BT como paucibacilares, segundo a Organização Mundial de Saúde (7).
IMUNIDADE INATA NA HANSENÍASE
Embora enorme atenção tenha sido focada no desenvolvimento da resposta imune celular adaptativa, durante o curso da infecção, investigações recentes dos mecanismos e modulação da imunidade inata suportam a ideia de que, após a forma Indeterminada de hanseníase, devem ocorrer eventos imunorregulatórios, os quais determinam o espectro da doença.
Células dendríticas (CDs), derivadas de células mononucleares sob a influência de IL(Interleucina)-4 e GM-CSF (Fator de crescimento de Granulócito e Macrófago) são apresentadoras muito efetivas de M. leprae (8). Atuando no sítio de invasão do bacilo, CDs podem ser as primeiras células a encontrá-lo e a exercerem uma resposta imune inata precoce contra o mesmo. Dependendo do nível da maturação das CDs, da interação com o bacilo e com componentes, tais como a membrana celular do M. leprae ou PGL (glicolipídeo-fenólico)-1, da subsequente produção local de citocinas pró e anti-inflamatórias (IL-2, IL-12, TNF-α, IFN-γ vs. IL-4, IL-10, TGF-β1) e de quimiocinas apropriadas, poderia haver regulação da inflamação e, consequentemente, respostas Th1 ou Th2 ao M. leprae.
A imunidade natural pode ter um papel no resultado da infecção, por meio da produção de citocinas que direcionam a resposta para o padrão produzido por células T. Macrófagos infectados liberam IFN-α e IL-12, os quais estimulam células Natural Killer (NK) a liberarem IFN-γ, colaborando na manutenção do padrão Th1 (9). A produção de IFN-γ favorece a manutenção da ativação de macrófagos e, consequentemente, a formação do granuloma imune (10).
IMUNIDADE ADAPTATIVA NA HANSENÍASE
A imunidade mediada por célula e humoral é dinâmica e apresenta variações espontâneas de reatividade com o tempo e tratamento, caracterizando as reações hansênicas agudas, tipo I e II.
Em infecções micobacterianas, a liberação de IL-2 e IFN-γ está, geralmente, associada com a resistência a infecções intracelulares, enquanto a liberação de IL-4 e IL-10 está associada com a doença progressiva. Pacientes paucibacilares do tipo HT e BT apresentam alta produção local (nas lesões) de citocinas do tipo Th1, como IFN-γ, IL-2, IL-7, IL-12, IL-15 e IL-18, características de resposta imune celular intensa. IFN-γ ativa macrófagos infectados, enquanto a IL-2 pode induzir a expansão clonal de células T ativadas e aumentar a produção de IFN-γ (11). A Figura 1 ilustra o padrão da liberação de citocinas na hanseníase.
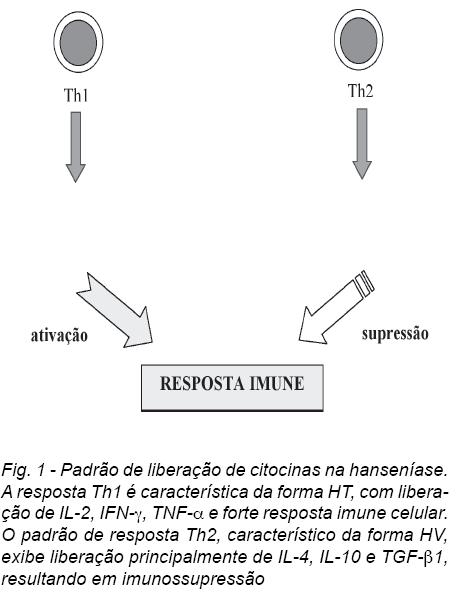
Indivíduos multibacilares possuem um padrão de citocinas do tipo Th2 nas lesões (IL-4, IL-5 e IL-10), indicando ineficaz resposta imune celular com alta produção de anticorpos. No entanto, estes anticorpos não são protetores da doença. A IL-4 tem um efeito imunorregulatório negativo sobre a imunidade mediada por células, o qual pode levar ao aumento da proliferação bacteriana, porque bloqueia a proliferação dependente de IL-2 de células T humanas porinibir receptores de IL-2; bloqueia a ativação de monócitos mediada pelo IFN-γ; inibe a expressão de CD14 sobre monócitos e produção de IL-1b e TNF-α; e, bloqueia a geração de óxido nítrico, necessário para a destruição de patógenos intracelulares (12).
Uma ativação crônica local de IL-10 pode levar a uma diferenciação de células T CD4+, originando uma subpopulação de células T regulatórias (Tr1) que produzem altos níveis de IL-10, mantendo a supressão da resposta imune antígeno-específica (13).
O TGF-β1 é produto, principalmente, de monócitos ativados, uma molécula bifuncional (pró-inflamatória e imunossupressora). Realiza supressão de linfócitos T – inibindo a expressão de IFN-γ e IL-2 e, possui a habilidade de inibir a citotoxicidade de macrófagos, permitindo a progressão da doença (14). Além da ação supressora, induz efeitos pró-inflamatórios na reação de tipo II em pacientes multibacilares (15).
Concluindo, o destino da infecção por M. leprae em um hospedeiro parece depender de quando e como uma determinada citocina está disponível no sítio e, da presença do parasita em maior quantidade em relação a vários outros produtos (16). Nesse contexto, deve estar inserida a predisposição genética do indivíduo na susceptibilidade ou resistência à infecção por M. leprae.
GENÉTICA DE SUSCEPTIBILIDADE
A incidência da infecção parece ser muito maior que a incidência de hanseníase clínica, pois somente uma pequena proporção dos infectados desenvolvem os sintomas clínicos (17). Isto pode ser devido, em parte, aos fatores ambientais, como a nutrição ou diferenças bacterianas genéticas, mas as evidências maiores são em relação à base genética de susceptibilidade do hospedeiro a ambos, à doença per se e aos subtipos da doença (Figura 2).
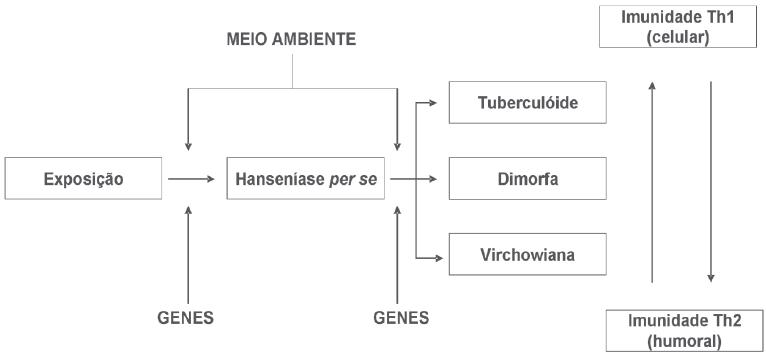
Fig. 2 - Modelo para o espectro da hanseníase de acordo com Prevedello e Mira (2007) (65). Após a exposição, a maioria dos indivíduos é resistente à hanseníase. Os susceptíveis podem apresentar a infecção per se ou desenvolver uma das formas clínicas do espectro da hanseníase, as quais são dependentes do padrão de resposta imune apresentada pelo hospedeiro.
Há muitos anos, as pesquisas buscam conhecer a genética de predisposição à hanseníase per se e a uma particular forma da doença. Numa revisão recente, Goulart e Goulart (2009) relataram algumas dessas associações genéticas com a hanseníase (18). O espectro clínico e patológico da hanseníase e a heterogeneidade epidemiológica, geográfica e étnica podem ser explicados pelas diferenças genéticas na resistência do hospedeiro. Enquanto alguns locos afetam a susceptibilidade intrínseca à hanseníase (hanseníase per se), outros modificam a forma clínica da doença (19). Análises complexas de segregação, em várias populações, relataram modelos consistentes com um gene principal codominante ou recessivo e, talvez, vários genes controlando a susceptibilidade à doença (20, 21).
A destruição ou a multiplicação do bacilo no interior dos macrófagos pode ser determinada por mecanismos imunológicos, os quais envolvem a apresentação de peptídeos bacterianos associados aos antígenos leucocitários humanos (HLA) aos linfócitos T (22). Moléculas HLA são codificadas pelo Complexo Principal de Histocompatibilidade (CPH), localizado no braço curto do cromossomo 6 humano, o qual é uma região candidata para controlar a susceptibilidade à hanseníase (23).
Além disso, vários estudos de associação apontaram o envolvimento de variantes HLA no controle da resposta imune ao bacilo, a qual é responsável pela evolução da doença às distintas formas clínicas (24-27). Segundo Mira et al. (2003), as variantes alélicas HLA podem ser fatores de risco para as formas pauci e multibacilares da hanseníase (28). No entanto, Vanderborght et al. (2007) encontraram associação entre o loco HLA-DRB1 e a hanseníase per se, mas não com os subtipos clínicos da doença, em duas populações: brasileira e vietnamita (29). Eles verificaram que HLA-DRB1*04 estava associado à proteção contra a hanseníase, enquanto o alelo HLA-DRB1*10 determinava a susceptibilidade. Na Argentina, Motta et al. (2007) também observaram uma associação de HLA-DRB1*04 com a proteção, mas somente contra a forma clínica paucibacilar da doença (30). Numa população do Paraná, um recente estudo confirmou a proteção exercida pelo alelo DRB1*04, porém em relação à forma mais grave da doença (HV). Ainda, apontou outros alelos de susceptibilidade à hanseníase per se: HLA-DRB1*16 e à forma HV: HLA-DRB1*08 (31).
Análises por meio de escaneamento de genoma com pacientes hansenianos identificaram um loco de susceptibilidade na região do cromossomo 10p13 na Índia (32) e outro, conduzido no Brasil, sugeriu um papel para as regiões 6p21 e 17q22 (33). Posteriormente, um estudo realizado em 2003 verificou uma associação da susceptibilidade à hanseníase per se com a região 6q25q26 em famílias vietnamitas (29). Mais tarde, em 2004 as variantes PARK2 e PACRG foram identificadas nesta região como sendo fortes fatores de susceptibilidade à hanseníase (34). Em populações do Vietnã, Índia e Brasil (35), o gene que codifica a linfotoxina alfa (LTA, na região 6p21 do MHC classe III) foi associado ao aumento da susceptibilidade à hanseníase per se.
Vários estudos de polimorfismos únicos de nucleotídeos (SNPs, do inglês, single nucleotide polymorphisms) foram realizados na busca de associações com doenças (36), pois esses são considerados a fonte mais abundante de variação no genoma humano. Quando presentes num gene podem provocar diferenças na expressão de proteínas, causando mudanças estruturais e funcionais. Alguns SNPs em genes de citocinas foram descritos como importantes fatores genéticos na ocorrência das distintas formas clínicas da hanseníase.
Pacientes com genótipos -819TT, referentes ao gene da IL10, foram, significativamente, associados com a hanseníase numa população brasileira do Rio de Janeiro (37). Outro estudo, em pacientes da mesma região definiu como marcador de resistência à hanseníase o haplótipo -3575A/-2849G/-2763C (38). Em pacientes indianos, a presença do haplótipo -3575T/-2849G/-2763C/-1082A/-819C/-592C conferiu resistência à hanseníase per se e o haplótipo-3575T/-2849G/-2763C/-1082A/-819T/-592A foi associado com o risco de desenvolver a forma mais grave da doença (39). Numa população do Paraná, nossa equipe mostrou que o haplótipo IL10-1082G/-819C/-592C estava diminuído em pacientes HV, em relação aos controles (40). Recentemente, outro estudo brasileiro confirmou a associação do alelo -819T com a susceptibilidade à hanseníase (41).
Em relação ao gene TNF que se encontra na região do CPH, a frequência do alelo TNF2 (com substituição G→A na posição -308 – região iniciadora da transcrição do TNF) foi encontrada, significativamente, aumentada em pacientes com a forma HV, em relação aos controles, com risco relativo de 2,5 numa população da Inglaterra (42). Este resultado foi semelhante ao encontrado num estudo tailandês que observou esse alelo associado a hansenianos multibacilares (43). No entanto, dois estudos independentes, realizados em populações brasileiras (37,40), indicaram associação do alelo A com a proteção ao desenvolvimento da doença. Considerando, que o papel biológico dos polimorfismos em regiões promotoras do gene TNF não está totalmente esclarecido (44), resultados controversos como estes podem surgir, sugerindo a participação do loco do TNF no intercurso da hanseníase. A interação entre polimorfismos, localizados em regiões promotoras deste gene, pode ser a responsável pelos efeitos distintos na produção desta citocina observados em algumas populações.
Por meio da genotipagem do gene IL12B, que codifica a subunidade p40 das citocinas IL-12 e IL-23, Morahan et al. (2007) encontraram que a modulação da produção de IL-12p40 exerce influência na hanseníase e na tuberculose (45). O grau de expressão dos genes das subunidades do receptor da IL-12 (IL12RB1 e IL12RB2) também foi testado. A frequência do haplótipo -1035A/-1023A/-650G/-464A na região flanqueadora da extremidade 5’ do gene IL12RB2 mostrou-se diminuída em pacientes HV do Japão, em relação aos HT e aos controles (46). Em pacientes coreanos, SNPs do gene IL12RB1 não foram associados com a doença, assim como polimorfismos do IFNGR1 (receptor 1 do Interferon-γ) (47).
Outro candidato à susceptibilidade à hanseníase é o gene NRAMP1 (proteína 1 macrofágica associada à resistência natural), desde que seu homólogo murino foi associado à resistência contra o Mycobacterium lepraemurium (48). No estudo de Ferreira et al. (2004), foi encontrado que este gene exibe uma interação com a resposta à lepromina e, que um alelo do NRAMP1 (alelo 2) é fator de predisposição à doença (49). Esses dados confirmam os resultados de Alcais et al. (2000), num estudo com famílias com a doença no Vietnã (6).
Os TLRs (Toll-like Receptors) também apresentam SNPs que podem estar envolvidos na imunopatologia da doença. Formam uma família de receptores transmembrânicos que permitem ao hospedeiro reconhecer um grande número de padrões moleculares de microorganismos, como lipopolissacarídeos bacterianos, RNAs virais e a PGL-1 (50). Kang e Chae (2001) observaram que pacientes HV possuem uma substituição C�T na posição 2029 do códon iniciador do gene TLR2, a qual poderia ocasionar uma mutação no domínio intracelular do receptor levando à susceptibilidade a essa forma (51). Outra substituição, de arginina por triptofano na posição 677 do TLR2, mostrou-se associada à forma HV numa população coreana (52). Mais recentemente, o TLR1 e TLR4 foram associados com a susceptibilidade de desenvolver a hanseníase e suas formas clínicas (53,54).
Certos genótipos da forma ativa da vitamina D podem estar relacionados à hanseníase. Além de regularem o metabolismo do cálcio, apresentam importante papel imunorregulatório através de sua ligação com receptores VDR (receptores de vitamina D) na superfície de monócitos, macrófagos e linfócitos (55). Sua sinergia com outros fatores genéticos também parece afetar a imunidade celular na doença (56). Um estudo na população mexicana mostrou que o genótipo TT do gene TaqI do receptor da vitamina D estava associado com a forma HV da doença (57).
Outros genes de resposta imune foram estudados no intuito de esclarecer sua possível participação na ocorrência ou gravidade de uma doença. Dentre eles, podemos destacar os genes MICA e MICB (do inglês, Major Histocompatibility Complex class I chain-related genes A and B). Estes genes se encontram no cromossomo 6 (58), próximos aos HLA-B e -C e, devido ao seu alto grau de polimorfismo, variantes alélicas poderiam gerar distintas proteínas MICA induzidas pelo estresse. Proteínas MICA são conhecidas por servirem como ligantes para o receptor NKG2D de ativação da célula NK, o qual é também expresso na superfície de células T CD8+ (59). Estas poderiam se ligar ao receptor NKG2D de forma diferente, afetando a imunopatogênese de uma doença. Polimorfismos nestes genes foram investigados quanto ao seu papel nas doenças infecciosas (60) e, um estudo de casos-controles no Sul da China, sugeriu que uma variante do éxon 5 do gene MICA poderia estar associada com a resistência à forma multibacilar da doença (61). Na Índia, o alelo MICA*5A5.1 foi associado à susceptibilidade à hanseníase per se (62).
Recentemente, os genes KIR (do inglês Killer-cell Immunoglobulin-like Receptor) que codificam receptores inibitórios e estimulatórios de células NK foram estudados, pela primeira vez, em pacientes brasileiros com hanseníase e mostraram influenciar na evolução clínica da doença (63). Estes receptores se ligam às moléculas HLA de classe I e reconhecem as células-alvo para sua destruição ou não. Avariação de sequências em KIR pode ocorrer em posições que codificam resíduos que afetam a interação com moléculas ligantes HLA de classe I (64). Quando ligados, os KIR inibitórios protegem as células da eliminação por células NK. No estudo de Franceschi et al. (2008), genes de receptores ativatórios, KIR2DS2 e KIR2DS3, foram encontrados em maior frequência no grupo de pacientes HT em relação ao grupo HV nesta população, o que poderia determinar uma melhor resposta de citotoxicidade da célula NK frente ao bacilo neste grupo com HT (63). Também foram demonstradas diferenças nas frequências de genes KIR inibitórios e seus ligantes entre os subgrupos. As frequências de KIR2DL1 e de seu ligante C2 (HLA-C*02, *04, *05, *06, *07, *15, *17 e *18) foram menores no grupo HD, comparadas aquelas vistas nos controles e pacientes HT. Tal fato reforça a questão de instabilidade da resposta imune frente ao M. leprae nesta forma da doença. Além disso, foi observada uma frequência reduzida de KIR2DL3 com C1 (HLA-C*01, *03, *07, *08, *12, *13, *14 e *16), em pacientes HT, quando comparada aos controles, aos pacientes com hanseníase per se e ao grupo HD. Este fato pode contribuir para a melhor resposta imune celular característica de pacientes HT.
CONCLUSÃO
Em resumo, a hanseníase é uma doença multifatorial complexa, pois o desenvolvimento da infecção e das formas clínicas, após o contato com o bacilo, está sob o controle de fatores físicos e ambientais, além, de fatores genéticos do hospedeiro. No entanto, podemos concluir que, a genética do hospedeiro exerce uma importante função em determinar a ocorrência da infecção e sua evolução clínica e imunológica. Daí, a importância em identificá-los numa população única, para avaliarmos se a presença de um ou mais destes fatores de risco, num indivíduoinfectado, poderia contribuir para o aparecimento das manifestações clínicas.
PERSPECTIVAS E AÇÕES FUTURAS
Novos estudos, envolvendo populações de distintas partes do mundo e fatores genéticos já descritos para a hanseníase naquelas regiões, devem ser conduzidos para avaliar a interação entre estes fatores. Somente desta forma, poderemos chegar a uma definição mais clara destas associações genéticas e das interações entre estas proteínas, as quais muitas vezes ocorrem resultando numa resposta imune eficaz ou não a um agente infeccioso.
Uma perspectiva é o desenvolvimento de estudos de associação genética envolvendo as reações agudas que se manifestam em alguns pacientes hansenianos. Estas representam a reativação da respostaimune celular associada à produção de várias citocinas inflamatórias. Estas reações podem resultar em sérios danos à saúde destes pacientes, pois causamlesões teciduais e neurais, ocasionando deformidades que aumentam o estigma da doença. Uma vez que tais reações pudessem ser previstas, haveria a possibilidade de preveni-las, pela escolha de uma terapêutica mais adequada, proporcionando uma melhor qualidade de vida a estes indivíduos.
Nesta nova Era pós-genômica, a possibilidade da realização de estudos com um número grande de marcadores genéticos da hanseníase numa única população, por meio de análises de polimorfismos de genes com plataformas baseadas em Microarrays, poderá permitir a definição de um perfil genético de risco característico desta patologia.
Agradecimentos
Os autores agradecem àqueles que colaboraram na correção do manuscrito.
REFERÊNCIAS
1 -World Health Organization. Globalleprosy situation. Weekly Epidemiological Record 2008;83:293-300.
2 -Ministério da Saúde. Secretaria de Vigilância em Saúde. Programa Nacional de eliminação da hanseníase. Disponível em: http://portal.saude.gov.br/portal/saude/visualizar_texto.cfm?idtxt=27640. Acesso em: 17/06/2009.
3 -Alter A, Alcaïs A, Abel L, Schurr E. Leprosy as a genetic model for susceptibility to common infectious diseases. Hum Genet 2008;123:227-35.
4 -Cole ST, Eiglmeier K, Parkhill J, James KD, Thomson NR, Wheeler PR, et al. Massive gene decay in the leprosy bacillus. Nature 2001;409:1007-11.
5 -Ridley DS, Jopling WH. Classification of leprosy according to immunity. A five-group system. Int Lepr Other Mycobac Dis 1966;34:255-73.
6 -Alcais A, Sanchez FO, Thuc NV, et al. Granulomatous reaction tointradermalinjection oflepromin (Mitsuda Reaction) is linked to the human NRAMP1 gene in Vietnamese leprosy sibships. J Infect Dis 2000;181:302-8.
7 -World Health Organization. Chemotherapy of leprosy for control programs. WHO, Tech Rep Ser 1982;675:1-33.
8 -Sieling PA, Jullien D, Dahlem M, et al. CD1 expression by dendritic cells in human leprosy lesions: Correlation with effective host immunity. J Immunol 1999;162:1851-58.
9 -Goulart IM, Penna GO, Cunha G. Immunopathology of leprosy: the complexity of the mechanisms of host immune response to Mycobacterium leprae. Rev Soc Bras Med Trop 2002;35:365-75.
10 -Silva CL, Foss NT. Tumor necrosis factorinleprosy patients. J Infec Dis 1989;159:787-90.
[ Links ]11 -Kasahara T, Hooks JJ, Dougherty SF, Oppenheim JJ. Interleukin 2-mediated immune interferon (IFN-gamma) production by human T cells and T cell subsets. J Immunol 1983;130:1784-89.
12 -Sieling PA, Modlin RL. Cytokine patterns at the site of mycobacterial infection. Immunobiol 1994;191:378-87.
13 -Asseman C, Powrie F. Interleukin 10 is a growth factor for a population of regulatory T cells. Gut 1998;42:157-8.
14 -Kiszewski CA, Becerril E, Baquera J, Aguilar LD, Hernán-dez-Pando R. Expression of transforming growth factor-beta isoforms and their receptorsinlepromatous and tuberculoid leprosy. Scand J Immunol 2003;57:279-85.
15 -Goulart IM, Mineo JR, Foss NT. Production of transforming growth factor-beta 1 (TGF-beta1) by blood monocytes from patients with different clinical forms of leprosy. Clin Exp Immunol 2000;122:330-4.
16 -Goulart IM, Figueiredo F, Coimbra T, Foss NT. Detection of transforming growth factor-b1 in dermal lesions of different clinical forms of leprosy. Am J Pathol 1996;148:911-7.
17 -Fine PEM. Natural history of leprosy – Aspects relevant to a leprosy vaccine. Int J Leprosy 1983;51:553-5.
18 -Goulart LR, Goulart IMB. Leprosy pathogenetic background: a review and lessons from other mycobacterial diseases. Arch Dermatol Res 2009;301:123-37.
19 -Schurr E, Alcaïs A, de Léséleuc L, Abel L. Genetic predisposition to leprosy: A major gene reveals novel pathways of immunity to Mycobacterium leprae. Semin Immunol 2006;18:404-10.
20 -Abel L, Demenais F. Detection of major genes for susceptibility to leprosy and its subtypes in a Caribbean island: Desirade island. Am J Hum Genet 1988;42:256-66.
21 -Feitosa MF, Borecki I, Krieger H, Beiguelman B, Rao DC. The genetic epidemiology of leprosy in a Brazilian population. Am J Hum Genet 1995;56:1179-85.
22 -Foss NT. Hanseníase: aspectos clínicos, imunológicos e terapêuticos. An Bras Dermatol 1999;74:113-9.
23 -Dessoukey MW, El Shiemy S, Sallam T. HLA and leprosy: Segregation and linkage study. Int J Dermatol 1996; 35:257-64.
24 -Visentainer JEL, Tsuneto LT, Serra MF, Peixoto PRF, Petzl-Eler ML.Association ofleprosy with HLA-DR2in a Southern Brazilian population. Braz J Med Biol Res 1997;30:51-9.
25 -Meyer CG, May J, Stark K. Human leukocyte antigens in tuberculosis and leprosy. Trends in Microbiol 1998; 6:148-54.
26 -Hegazy AA, Abdel-Hamid IA, Ahmed el-SF, Hammad SM, Hawas SA. Leprosy in a high-prevalence Egyptian village: epidemiology and risk factors. Int J Dermatol 2002; 41:681-6.
27 -Gorodezky C, Flores J, Arevalo N, Castro LE, Silva A, Rodriguez O. Tuberculoid leprosy in Mexicans is associated with HlLA-DR3. Lepr Rev 1987;58:401-6.
28 -Mira MT, Alcais A, di Pietrantonio T, et al. Segregation of HLA/TNF region is linked to leprosy clinical spectrum in families displaying mixed leprosy subtypes. Genes Immun 2003;4:67-73.
29 -Vanderborght PR, Pacheco AG, Moraes ME, et al. HLA-DRB1*04 and DRB1*10 are associated with resistance and susceptibility, respectively, in Brazilian and Vietnamese leprosy patients. Genes Immun 2007;8:320-4.
30 -Motta PM, Cech N, Fontan C, et al. Role of HLA-DR and HLA-DQ alleles in multibacillary leprosy and paucibacillary leprosy in the province of Chaco (Argentina). Enferm Infecc Microbiol Clin 2007;25:627-31.
31 -Silva SA, Mazini PS, Reis PG, et al. HLA-DR and HLADQ alleles in patients from the south of Brazil: markers for leprosy susceptibility and resistance. BMC Infect Dis 2009;9:134-40.
32 -Siddiqui MR, Meisner S, Tosh K, et al. A major susceptibility locus for leprosy in India maps to chromosome 10p13. Nat Genet 2001;27:439-41.
33 -Miller EN, Jamielson SE, Joberty C, et al. Genome-wide scans for leprosy and tuberculosis susceptibility genes in Brazilians. Genes Immun 2004; 5:63-7.
34 -Mira MT, Alcais A, Nguyen VT, et al. Susceptibility to leprosy is associated with PARK2 and PACRG. Nature 2004; 427:636-40.
35 -Alcais A, Alter A, Antoni G, et al. Stepwise replication identifies a low-producing lymphotoxin-a allele as a major risk factor for early-onset leprosy. Nat Genet 2007; 39:517-22.
36 -Suh Y, Vijg J. SNP discovery in associating genetic variation with human disease phenotypes. Mutat Res 2005; 573:41-53.
37 -Santos AR, Suffys PN, Vanderborght PR, Moraes MO, Vieira LMM, Cabello PH, et al. Role of Tumor Necrosis Factor–a and Interleukin-10 Promoter Gene Polymorphisms in Leprosy. J Infect Dis 2002;186:1687-91.
38 -Moraes MO, PachecoAG, Schonkeren JJ, et al. Interleukin10 promoter single-nucleotide polymorphisms as markers for disease susceptibility and disease severity in leprosy. Genes Immun 2004;5:592-5.
39 -Malhotra D, Darvishi K, Sood S, et al. IL-10 promoter single nucleotide polymorphisms are significantly associated with resistance to leprosy. Hum Genet 2005;118:295-300.
40 -Franceschi DS, Mazini PS, Rudnick CC, et al. Influence of TNF and IL10 gene polymorphisms in the immunopathogenesis of leprosy in the South of Brazil. Int J Infect Dis 2009;13:493-8.
41 -Pereira AC, Brito-de-Souza VN, Cardoso CC, et al. Genetic, epidemiological and biological analysis of interleukin-10 promoter single-nucleotide polymorphisms suggests a definitive role for -819C/T in leprosy susceptibility. Genes Immun 2009;10:174-80.
42 -Roy S, McGuire W, Mascie-Taylor CGN, Hazra SK, Hill AVS, Kwiatkowski D. Tumor Necrosis Factor Promoter Polymorphism and Susceptibility to Lepromatous Leprosy. J Infect Dis 1997;176:530-2.
43 -Vejbaesya S, Mahaisavariya P, Luangtrakool P, Sermduangprateep C. TNF alpha and NRAMP1 polymorphisms in leprosy. J Med Assoc Thai 2007;90:1188-92.
44 -Bayley JP, Ottenhoff THM, Verweij CL. Is there a future for TNF promoter polymorphisms? Genes Immun 2004; 5:315-29.
45 -Morahan G, Kaur G, Singh M, et al. Association of variants in the IL12B gene with leprosy and tuberculosis. Tissue Antigens 2007;69:234-6.
46 -Ohyama H, Ogata K, Takeuchi K, Namisato M, Fukutomi Y, Nishimura F. Polymorphism of the 5’ flanking region of the IL-12 receptor ß2 gene partially determines the clinical types of leprosy through impaired transcriptional activity. J Clin Pathol 2005;58:740-3.
47 -Lee SB, Kim BC, Jin SH, Park YG, et al. Missense mutations of the interleukin-12 receptor beta 1(IL12RB1) and interferon-gamma receptor 1(IFNGR1) genes are not associated with susceptibility to lepromatous leprosy in Korea. Immunogenetics 2003;55:177-81.
48 -Abel L, Sánchez FO, Oberti J, et al. Susceptibility to leprosy is linked to the human NRAMP1 gene J Infect Dis. 1998;177:133-45.
49 -Ferreira FR, Goulart LR, Silva HD, Goulart IM. Susceptibility to leprosy may be conditioned by an interaction between the NRAMP1 promoter polymorphisms and the lepromin response. International Journal of Leprosy and Other Mycobacterial Diseases 2004;72:457-67.
50 -Chen K, Huang J, Gong W, Iribarren P, Dunlop NM, Wang JM. Toll-like receptorsininflammation,infection and cancer. Int Immunopharmacol 2007;7:1271-85.
51 -Kang TJ, Chae GT. Detection of Toll-like receptor 2 (TLR2) mutation in the lepromatous leprosy patients. FEMS Immunol Med Microbiol 2001;31:53-8.
52 -Malhotra D, Relhan V, Reddy BS, Bamezai R. TLR2 Arg677Trp polymorphism in leprosy: revisited. Hum Genet 2005;116:413-5.
53 -Bochud PY, Sinsimer D, Aderem A, et al. Polymorphisms in Toll-like receptor 4 (TLR4) are associated with protection against leprosy. Eur J Clin Microbiol Infect Dis 2009;28:1055-65.
54 -Schuring RP, Hamann L, Faber WR, et al. Polymorphism N248S in the human Toll-like receptor 1 gene is related to leprosy and leprosy reactions. J Infect Dis 2009;199:1816-9.
55 -Roy S, Frodsham A, Saha B, Hazra SK, Mascie-Taylor CGN, Hill AVS. Association of vitamin D receptor genotype with leprosy type. J Infec Dis 1999;179:187-91.
56 -Félix JS, Salazar SG, Velázquez RC, Maldonado JG, Villalobos HR. Relación del polimorfismo TaqI del gen del receptor de la vitamina D con la lepra lepromatosa en población Mexicana. Salud Publica Mex 2009;51:59-61.
57 -Goulart LR, Ferreira FR, Goulart IM. Interaction of TaqI polymorphism at exon 9 of the vitamin D receptor gene with the negative lepromin response may favor the occurrence of leprosy. FEMS Immunol Med Microbiol 2006;48:91-8.
58 -Bahram S, Bresnahan M, Geraghtyt DE, Spies T. A second lineage of mammalian major histocompatibility complex class I genes. Proc Natl Acad Sci USA 1994;91:6259-63.
59 -Hue S, Monteiro RC, Berrih-Aknin S, Caillat-Zucman S. Potential role of NKG2D/MHC class I-related chain A interaction in intrathymic maturation of single-positive CD8 T cells. J Immunol 2003;71:1909-17.
60 -González S, Rodriguez-Rodero S, Martinez-Borra J, López-Vázquez A, Rodrigo L, López-Larrea C. MICB typing by PCR amplification with sequence specific primers. Immunogenetics 2003;54:850-5.
61 -Wang LM, Kimura A, Satoh M, Mineshita S. HLA linked with leprosy in southern China: HLA-linked resistance alleles to leprosy. Int J Lepr Other Mycobact Dis 1999;67:403-8.
62 -Tosh K, Ravikumar M, Bell JT, Meisner S, Hill AV, Pitchappan R. Variation in MICA and MICB genes and enhanced susceptibility to paucibacillary leprosy in South India. Hum Mol Genet 2006;15:2880-7.
63 -Franceschi DS, Mazini PS, Rudnick CC, et al. Association between killer-cell immunoglobulin-like receptor (KIR) genotypes and leprosy in Brazil. Tissue Antigens 2008; 72:478-82.
64 -Boyington JC, Sun PD. A structural perspective on MHC class I recognition by killer cell immunoglobulin-like receptors. Mol Immunol 2002;38:1007-21.
65 -Prevedello FC, Mira MT. Hanseníase: uma doença genética? An Bras Dermatol 2007;82:451-9.
Correspondência:
Dr.ª Jeane Eliete Laguila Visentainer
Departamento de Ciências Básicas da Saúde
Universidade Estadual de Maringá
Av. Colombo, 5790
CEP: 87020-900 Maringá - PR - Brasil
e-mail: jelvisentainer@uem.br