Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Portuguesa de Imunoalergologia
versão impressa ISSN 0871-9721
Rev Port Imunoalergologia vol.21 no.4 Lisboa dez. 2013
ARTIGO DE REVISÃO
Imunogenética das reacções alérgicas a fármacos
Immunogenetics of allergic reactions to drugs
Natacha Santos, Josefina Rodrigues Cernadas
Serviço de Imunoalergologia, Centro Hospitalar São João, Porto
RESUMO
A base genética das reacções alérgicas a fármacos pode relacionar -se com genes nas vias de activação ou inactivação que afectam a farmacocinética ou farmacodinâmica, no reconhecimento e resposta do sistema imunológico (imunogenética) ou nos mecanismos de lesão e reparação tecidular. O objectivo deste trabalho é apresentar uma revisão actua lizada da imunogenética das reacções alérgicas a fármacos, de acordo com o provável mecanismo fisiopatológico envolvido, com particular ênfase na relação entre determinados polimorfismos do HLA e a alergia do tipo IV. Para além de uma melhor compreensão dos mecanismos imunológicos, esta é uma área com imediata aplicabilidade clínica, como no caso da prevenção primária de reacção de hipersensibilidade ao abacavir através do rastreio do HLA-B*5701 e a outros fármacos, como a carbamazepina nas populações asiáticas. Numa área em que, cada vez mais, a investigação assume um carácter de cooperação internacional, o papel do especialista em Imunoalergologia é essencial para o correcto diagnóstico dos doentes com alergias a fármacos e registo das reacções em bases nacionais e internacionais.
Palavras-chave: Alergia a fármacos, farmacogenética, hipersensibilidade a fármacos, imunogenética.
ABSTRACT
The genetic basis of drug allergy can be related to activation or inactivation pathways that affect the pharmacokinetics or pharmacodynamics, in the immune system recognition and response, or in the lesion and tissue repair mechanisms. The aim of this article is to present an up-to-date review of drug allergy immunogenetics according to the probable physiopathological mechanism involved, with particular emphasis on the relation between several HLA polymorphisms and type IV allergy. Besides a better understanding of the immunological mechanisms, this is a field with immediate clinical applicability, as in the primary prevention of abacavir-mediated hypersensitivity reactions through HLA -B*5701 screening, and of other drugs as carbamazepine in the Asian population. In a field where, more and more, investigation is performed with the need for international cooperation, the role of the Allergy and Clinical Immunology specialist is essential for the correct diagnosis of patients with drug allergy and registry in national and international databases.
Keywords : Drug allergy, drug hypersensitivity, immunogenetics, pharmacogenetics.
INTRODUÇÃO E DEFINIÇÕES
Alergia a fármacos
As reacções adversas a fármacos são definidas pela Organização Mundial de Saúde como uma resposta lesiva e não intencional a um fármaco, que ocorre em doses habitualmente utilizadas no homem1.
A classificação farmacológica clássica das reacções adversas a fármacos de Rawlins e Thompson divide as reacções em dois subtipos principais: reacções do tipo A, que são previsíveis e dose-dependentes, e reacções do tipo B, imprevisíveis e independentes da dose2. As reacções do tipo B são responsáveis por 10 a 15% de todas as reacções adversas a fármacos e incluem as reacções de hipersensibilidade.
De acordo com o Comité para a Revisão da Nomenclatura da Organização Mundial de Alergia, o termo hipersensibilidade deve ser utilizado para descrever reacções objectivamente reprodutíveis, iniciadas após a exposição a um estímulo definido e com uma dose habitualmente tolerada, enquanto o termo alergia a fármacos se refere a uma reacção de hipersensibilidade com mecanismo imunológico demonstrado3.
A alergia a fármacos é uma entidade heterogénea que compreende diferentes mecanismos imunológicos e manifestações clínicas, tal como proposto na classificação de Pichler (Figura 1)4. Foi também recentemente proposta por Pirmohamed et al.5 uma estandardização fenotípica das reacções cutâneas imunomediadas graves induzidas por fármacos (reacções alérgicas tipo IV) em: (i) Síndrome de Stevens-Johnson/necrólise epidérmica tóxica (SJS/TEN), (ii) pustulose exantemática generalizada aguda (AGEP), e (iii) síndrome de hipersensibilidade (HSS; também conhecido como reacção a fármacos com eosinofilia e sintomas sistémicos (DRESS) e síndrome de hipersensibilidade induzida por fármacos (DIHS)).
Farmacogenética e imunogenética
O risco de sensibilização e a gravidade do quadro clínico dependem do estado de activação imunológica do indivíduo, de cofactores como as infecções (ex.: infecção por citomegalovírus ou vírus herpes6, exantema com amoxicilina induzido pelo vírus Ebstein-Barr6 e aumento da hipersensibilidade ao trimetoprim-sulfameto xazole na infecção pelo vírus da imunodeficiência humana7), bem como de outros factores, como o género feminino, dose e duração do tratamento, via de administração (a aplicação epicutânea tem maior probabilidade de sensibilização do que os tratamentos orais ou parentéricos) e predisposição genética4.
O estudo da predisposição genética para reacções adversas a fármacos pode ser direccionado para o ADN (farmacogenética), o ARN (farmacotranscriptómica), a produção proteica e modificações pós-transcripcionais (farmacotranscriptómica) ou mesmo para o pool e interacção entre diferentes metabolitos (farmacometabolómica).
A farmacogenómica é um termo frequentemente usado indistintamente da farmacogenética, embora se refira ao estudo simultâneo de todos os genes (genoma) num grupo de indivíduos8.
A base genética das reacções alérgicas a fármacos pode relacionar-se com genes nas vias de activação ou inactivação que afectam a farmacocinética ou a farmacodinâmica, no reconhecimento e resposta do sistema imunológico ou nos mecanismos de lesão e reparação tecidular9.
Apesar das intensas investigações de polimorfismos em genes de metabolização dos fármacos e do seu papel na fisiopatologia das reacções de hipersensibilidade10-13, descobertas recentes têm focado a nossa atenção nos genes relacionados com o sistema imunológico (imunogenética) e a sua relação com a alergia a fármacos.
A maioria destes estudos foi realizada por uma abordagem que investiga um gene candidato ou uma via de activação previamente conhecida. No entanto, desde a sequenciação do genoma humano, em 2003, começaram a ser implementadas novas abordagens através de estudos de associação em todo o genoma – genome - wide association studies (GWAS) utilizando tecnologia de microarray para pesquisa de polimorfismos de um único nucleótido – single nucleotide polymorphisms (SNPs), o que irá certamente acrescentar um novo conhecimento neste tema.
O objectivo deste trabalho é apresentar uma revisão actualizada da imunogenética das reacções alérgicas a fármacos e dos possíveis mecanismos que nos são revelados através destas novas abordagens.
MATERIAL E MÉTODOS
Foi realizada pesquisa na Pubmed e Scopus utilizando as palavras -chave [(pharmacogenetics OR pharmacogenomics OR immunogenetics) AND (allergy OR hypersensitivity)] para artigos a partir de 1990 e com o limite human. A última pesquisa foi feita a 16 de Novembro de 2013 e identificou 1351 citações. Após ler o título e/ou abstract foram excluídos 1056 artigos por não se referirem ao objectivo da pesquisa. Foi ainda avaliada a bibliografia de recentes revisões sobre o tópico e pesquisa adicional sobre fármacos com informação insuficiente e adicionados os artigos não previamente incluídos. Foram encontrados 236 artigos de revisão, comentários ou editoriais relacionados com a farmacogenética, farmacoeconomia, medicina personalizada, reacções adversas a fármacos ou mecanismos imunológicos que incluem conceitos da imunogenética das reacções alérgicas a fármacos e que atestam o interesse e a importância deste tópico (Figura 2).
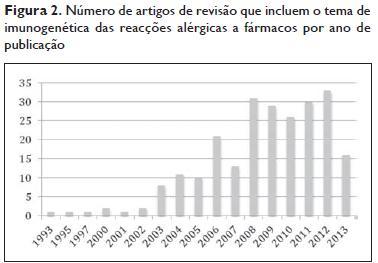
Foram também encontrados 160 artigos de investigação, de entre os quais seleccionados os artigos em texto completo ou em forma de resumo, sempre que este contivesse informação suficiente sobre o gene, população e fármaco avaliados, e tendo em conta as associações mais prevalentes ou de maior significado. Os resultados são apresentados de acordo com o provável mecanismo imunológico subjacente. Paralelamente aos artigos originais, são apresentados dados que sustentam o mecanismo fisiopatológico das alterações imunogenéticas encontradas, particularmente em relação com os polimorfismos do HLA.
RESULTADOS
Alergia tipo I
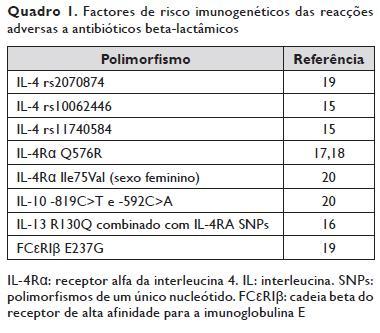
Os factores de risco para alergia IgE-mediada a fármacos podem envolver genes da apresentação antigénica, mudança de classe (class -switch) para IgE, expressão de receptores para a IgE (FCεR) ou das vias subsequentes de ativação e libertação de mediadores14, pelo que os estudos de farmacogenética têm avaliado a associação de alergia IgE -mediada a fármacos com polimorfismos nestas citocinas e receptores, especialmente na alergia a antibióticos beta -lactâmicos (Quadro 1). Os polimorfismos com maior significado foram reportados para as interleucinas IL-4 e IL-13, que influenciam a mudança de classe para IgE, bem como do receptor alfa da IL-4 (IL -4Rα) e receptor de alta afinidade para a IgE (FCεRI)15-19. Foi ainda reportada associação com polimorfismos da região promotora da IL -1020, que resultam na diminuição da síntese desta citocina com importante função inibitória de células pró-inflamatórias.
Também a interacção entre genes do CD40 e CD40 ligando foram associados a uma maior frequência de reacções alérgicas cutâneas (urticária, angioedema, exantema maculopapular e eritema multiforme) com vários antibióticos, incluindo penicilina, cefalosporinas e quinolonas, numa população coreana21.
A atopia, que é definida como a predisposição genética para gerar uma resposta IgE a moléculas inócuas, não está habitualmente associada a um maior risco de hipersensibilidade a fármacos. No entanto, pode prolongar a presença da IgE sérica específica para um determinado fármaco e a inflamação alérgica IgE -mediada pode agravar os sintomas de uma reacção alérgica IgE -mediada a fármacos4.
Alergia tipo II
Não foram encontrados estudos de imunogenética com este tipo de reacções.
Alergia tipo III
Foi descrita uma associação da deficiência em N -acetiltransferase tipo 2 (NAT2) – acetiladores lentos22 e de um polimorfismo de HLA -DR423 com o desenvolvimento de lúpus eritematoso sistémico induzido pela hidralazina.
No entanto, embora cada gene contribua para o risco de desenvolvimento da reacção alérgica, não são necessários nem suficientes para a sua ocorrência24.
Alergia tipo IV
No Quadro 2 são apresentados os polimorfismos HLA associados a diferentes fármacos e manifestações específicas, sendo os mais bem estudados o abacavir, a carbamazepina e o alopurinol, que se descrevem de seguida.
Abacavir
O abacavir é um análogo de nucleosídeo inibidor da transcriptase reversa, utilizado como medicamento de primeira linha no tratamento do vírus da imunodeficiência humana (VIH). Estudos prévios descreviam que 5 a 8% dos doentes sob tratamento com abacavir desenvolviam uma síndrome de hipersensibilidade induzida por fármacos (DIHS) caracterizada por febre, exantema generalizado não bolhoso, sintomas gastrintestinais e/ou envolvimento respiratório superior, até 6 semanas após o início do tratamento com abacavir (mediana de 7 a 10 dias)25.
A primeira evidência para uma base imunológica da DIHS com abacavir foi a demonstração histológica de células T CD8+ no exantema, a indução do factor de necrose tumoral alfa (TNFα) e interferão gama (IFNγ) no soro e/ou células mononucleadas e a presença de testes epicutâneos positivos26.
Em 2002 foi descrita por dois investigadores independentes27,28 uma forte associação entre a presença do alelo HLA-B*5701 e a DIHS ao abacavir. Estudos subsequentes em populações caucasianas e hispânicas confirmaram a força desta associação29,30. Em 2006 foi realizado um ensaio clínico prospectivo, randomizado e em dupla ocultação para avaliar o rastreio de ADN (estudo PREDICT), que envolveu 1956 participantes de 165 centros e 19 países e que demonstrou que o rastreio do HLA -B*5701 antes da introdução de abacavir exclui as DIHS imunologicamente confirmadas a este fármaco (valor preditivo negativo de 100%)31.
Subsequentemente, o estudo SHAPE conclui que, embora as DIHS imunologicamente confirmadas ao abacavir sejam pouco frequentes na raça negra, o alelo HLA-B*5701 é um marcador de DIHS tanto na população negra como na caucasiana nos Estados Unidos da América32.
Tendo em conta estes dados, a Food and Drug Adminstration (FDA)33, a Agência Médica Europeia (EMA)34 e a Sociedade Internacional de SIDA35, emitiram em 2008 recomendações no sentido de rastrear todos os doentes VIH positivos para o alelo HLA -B*5701 antes de iniciar tratamento com abacavir e optar por um anti–retrovírico alternativo nos doentes positivos para este alelo. Estudos em diferentes populações36,37, incluindo em Portugal38, confirmaram uma prevalência do gene HLA -B*5701 de 3,7 -6,1% para a maioria das populações, embora com prevalências inferiores a 2% para chineses39 e ugandeses40.
Para além disso, vários estudos de farmacoeconomia confirmaram o benefício desta abordagem nos Estados Unidos41, Alemanha42 e Espanha43, actualmente implementada na prática clínica. Salienta-se que foi também encontrada uma associação entre o HLA*B5701 e a lesão hepática causada pela flucloxacilina.
Carbamazepina
Em 2004 foi publicada a primeira associação genética forte entre a síndrome de Stevens -Johnson/necrólise epidérmica tóxica (SJS/TEN) induzida pela carbamazepina e o HLA -B*1502 em chineses han de uma coorte de Taiwan, com uma prevalência deste alelo em 100% dos 44 doentes, em comparação com apenas 3% dos 101 que toleraram a carbamazepina44. Este achado foi posteriormente confirmado em coortes de chineses residentes na Europa45 e Hong Kong46 e em outras populações asiáticas incluindo a tailandesa47, a indiana48 e a malaia49, motivando o alerta da FDA em 2007 no sentido de rastrear todos os doentes com antecedentes étnicos asiáticos, incluindo da Índia, para o HLA -B*1502 antes do início do tratamento com carbamazepina, optando por um antiepiléptico alternativo caso este alelo esteja presente50. Esta abordagem provou ser custo -efectiva na população chinesa e malaia de Singapura, mas não para a população de Singapura de etnia indiana51.
Pelo contrário, não foi encontrada associação entre o HLA -B*1502 e a SJS/TEN na população caucasiana52, nem em outras populações asiáticas, como a coreana53 ou a japonesa54, o que pode ser explicado em parte pela baixa frequência deste alelo nestes grupos étnicos, sendo que o HLA -B*1511 e o HLA -A*3101 foram os alelos com maior associação na Coreia53 e no Japão55, respectivamente. Por outro lado, não foi encontrada associação do alelo HLA-B*1502 com exantema maculopapular ou com DIHS em chineses, nos quais foi observada uma maior frequência de HLA -A*3101 e de polimorfismos específicos no gene da motilina, respectivamente56, o que indica que a associação de determinados polimorfismos a reacções alérgicas a um fármaco é provavelmente específica do fenótipo.
Nos caucasianos, Alfirevic et al.24,27 reportaram um potencial efeito protector do HLA-B*070252 e de três SNP do cluster do gene HSP-7057, na SJS/TEN induzida pela carbamazepina.
Alopurinol
A associação entre a SJS/TEN induzida pelo alopurinol e o HLA-B*5801 na população de chineses han foi publicada pela primeira vez em 200558. Esta associação foi posteriormente replicada em outras populações asiáticas incluindo a japonesa54, a tailandesa59, a coreana60 e também em caucasianos45,61, embora estatisticamente mais significativo para os chineses han (OR=580,3) do que para os europeus (OR=80)62. Tendo em conta estes dados, o departamento de saúde chinês actualizou a bula do alopurinol com a recomendação de testar o HLA-B*5801 antes de iniciar tratamento com alopurinol63, recomendações estas ainda não replicadas pela FDA ou pela EMA.
HLA e alergia tipo IV
O complexo major de histocompatibilidade (MHC) ou complexo antigénico dos leucócitos (HLA) nos humanos, é uma colecção de genes altamente polimórficos presentes no cromossoma 6. A molécula MHC da classe I é composta por uma glicoproteína com uma cadeia alfa pesada, codificada nos loci A, B e C do gene MHC da classe I, ligada à beta -2 -microglobulina. É expressa na superfície de praticamente todas as células nucleadas e a sua função principal é a apresentação de antigénios péptidos intracelulares endógenos às células T CD8+. A molécula MHC classe II é composta por uma cadeia de glicoproteína alfa e beta, codificadas nos loci DP, DQ e DR do gene MHC classe II. É expressa sobretudo nas células apresentadoras de antigénio (APC), onde apresenta péptidos exógenos processados às células T CD4+64.
A hipótese historicamente mais relevante para a maioria das reacções alérgicas a fármacos baseia-se na formação do complexo hapteno-péptido, que se liga ao MHC classe I ou II e é apresentado às células T65. É também actualmente proposta a hipótese do Perigo – Danger hypothesis, em que a toxicidade ou resposta inflamatória do próprio fármaco ou metabolitos induz a produção de moléculas coestimulatórias pelas células apresentadoras de antigénios (B7 -CD28, CD154 -CD40) necessárias à activação das células T naive após ligação do complexo hapteno-péptido/MHC ao receptor da célula T66.
No entanto, a mais recente hipótese para a patogénese da SJS/TEN baseia -se no conceito fármaco-imune – p -i concept, onde a apresentação às células T é dependente do HLA, directa, não covalente e farmacológica, levando à estimulação de células T efectoras e de memória sem necessidade de sensibilização prévia4,65, sendo este o modelo proposto para as reacções alérgicas associadas a polimorfismos específicos do HLA. Chessman et al. demonstraram que o reconhecimento do abacavir é feito pela via convencional de apresentação antigénica pelo MHC -II, com uma particular especificidade pelo HLA-B*570126. Os polimorfismos do HLA alteram a forma e a electroquímica das bolsas da fenda de ligação ao péptido (bolsas A, B, C, D, E e F no HLA classe I e bolsas P1, P4, P6, P7 e P9 no HLA classe II), afectando o reportório de péptidos que se podem ligar e ser apresentados por esse HLA67. As mutações que distinguem o HLA-B*5701 dos outros alelos estão predominantemente localizadas na bolsa F da fenda de ligação do antigénio, restringindo a ligação do abacavir a este alelo e não a outros alótipos relacionados como o HLA -B*570326.
Reacções de hipersensibilidade aos anti -inflamatórios não esteróides (AINE) As reacções de hipersensibilidade aos AINE representam um problema frequente para o imunoalergologista, com uma prevalência de 0,6 -0,9% na população geral, 2 -22% dos asmáticos e 21 -30% dos doentes com urticária crónica68. As reacções de hipersensibilidade aos AINE podem ser classificadas de acordo com a manifestação, reactividade cruzada, patologia e possível mecanismo subjacente, em (i) doença respiratória exacerbada pela aspirina – aspirin -exacerbated respiratory disease (AERD), (ii) doença cutânea exacerbada pela aspirina – aspirin-exacerbated cutaneous disease (AECD), (iii) urticária/angioedema induzido por múltiplos AINE – multiple non-steroid anti -inflamatory drugs -induced urticaria/angioedema (MNSAID -UA), (iv) alergia IgE -mediada a um único AINE, e (v) reacção por células T a um único AINE, conforme proposto por Ayuso et al.69, sendo que a maioria dos estudos farmacogenéticos tem incidido principalmente nos fenótipos de AERD, AECD e MNSAID-UD.
Apesar de terem sido encontradas associações com determinados alelos do HLA, como o HLA -DPB1*0301 na AERD70-72 e os HLA-DRB1*1302 e HLA -DQB1*0609 com urticária induzida ou exacerbada pelos AINE (AECD//MNSAID -UA)73 e HLA -DQ7 com alergia IgE -mediada a pirazolonas74, o valor preditivo destes está longe do encontrado para o abacavir ou a carbamazepina.
A maior parte dos polimorfismos encontrados estão relacionados com a via do ácido araquidónico e a produção de cisteinil leucotrienos (CysLT). Relativamente à AERD, foram encontradas associações com polimorfismos de genes das enzimas 5 lipoxigenase (ALOX-5) e leucotrieno C4 sintetase (LTC4S), importantes na síntese dos CysLT, na enzima N -acetiltransferase 2 (NAT2), que inativa os CysLT, e em receptores dos CysLT (CYSLTR1 e CYSLT2)71,72, bem como na enzima sintase 1 do tromboxano A (TBXAS1), no receptor do tromboxano A2 (TBXA2R), em receptores das prostaglandinas E e I (PTGER2, PTGER3, PTGER4 e PTGIR)71,72, no receptor CRTH2, responsável pela quimiotaxia de eosinófilos mediada pela prostaglandina D272,75 e no gene da subunidade gama 6 do canal de cálcio dependente de voltagem (CACNG6), uma proteína de membrana que estabiliza os canais de cálcio, prevenindo a hiperprodução de leucotrienos e recrutamento de outras células do sistema imunológico76. Nos fenótipos de MNSAID-UA foi também encontrada uma associação com polimorfismos da LTC4S77, CYSLTR178 TBXA2R79 e receptor da prostaglandina D (PTGDR)78, e um aparente papel protector de um polimorfismo da enzima lipoxigenase 15 (ALOX -15)78.
Foram também descritos polimorfismos na região promotora dos receptores de alta afinidade da IgE (FCεR1γ e FCεR1α), associados respectivamente a AERD71,72,80 e a AECD81. Outro polimorfismo descrito é o da histamina N -metiltransferase (HNMT), que diminui a actividade desta enzima, contribuindo para o aumento dos níveis de histamina e para o desenvolvimento de AECD82.
DISCUSSÃO E CONCLUSÃO
Como imunoalergologistas, devemos ter um papel activo junto das várias especialidades na melhoria do reconhecimento, caracterização fenotípica e correcta abordagem da alergia a fármacos. A utilização de estudos imunológicos como a IgE específica, testes cutâneos por picada, intradérmicos e epicutâneos, testes de transformação linfocitária e de activação dos basófilos devem ser utilizados, sempre que possível, de acordo com metodologias estandardizadas propostas em várias directrizes da Academia Europeia de Alergologia e Imunologia Clínica (EAACI) e da Rede Europeia de Alergia a Fármacos (ENDA). Por outro lado, é cada vez de maior importância a referenciação de informação completa e correcta para bases de dados internacionais, como a Drug Allergy and Hypersensitivity Database (DAHD)83, permitindo a melhoria do poder estatístico de estudos epidemiológicos relativamente aos factores genéticos que contribuem ou determinam as reacções alérgicas a fármacos. Os estudos farmacogenéticos têm-nos permitido compreender e conhecer melhor os mecanismos, melhorar o diagnóstico e até, como no caso do abacavir, prevenir as reacções alérgicas a fármacos. No futuro, esperam-se novos desenvolvimentos provenientes de estudos de associação de todo o genoma – genome -wide association studies (GWAS), bem como de outras áreas emergentes, como a farmacotranscriptómica, a metabolómica e a epigenética, que irão ajudar a melhorar a eficácia e segurança dos fármacos e guiar os clínicos na nova era da prescrição personalizada.
REFERÊNCIAS
1. International drug monitoring: the role of national centres. Report of a WHO meeting. World Health Organ Tech Rep Ser 1972;498:1-25. [ Links ]
2. Rawlins MD, Thompson JW. Pathogenesis of adverse drug reactions. In: Davies DM (Ed). Textbook of adverse drug reaction. Oxford: Oxford University Press; 1977:10-45. [ Links ]
3. Johansson SG, Bieber T, Dahl R, Friedmann PS, Lanier BQ, Lockey RF, et al. Revised nomenclature for allergy for global use: Report of the Nomenclature Review Committee of the World Allergy Organization, October 2003. J Allergy Clin Immunol 2004;113:832-6. [ Links ]
4. Pichler WJ. Drug hypersensitivity. Pichler WJ (Ed). Basel: Karger; 2007:168-89. [ Links ]
5. Pirmohamed M, Friedmann PS, Molokhia M, Loke YK, Smith C, Phillips E, et al. Phenotype standardization for immune–mediated drug-induced skin injury. Clin Pharmacol Ther 2011;89:896-901. [ Links ]
6. van der Linden PD, van der Lei J, Vlug AE, Stricker BH. Skin reactions to antibacterial agents in general practice. J Clin Epidemiol 1998;51:703-8. [ Links ]
7. Koopmans PP, van der Ven AJ, Vree TB, van der Meer JW. Pathogenesis of hypersensitivity reactions to drugs in patients with HIV infection: Allergic or toxic? AIDS 1995;9:217-22. [ Links ]
8. Kalow W. Pharmacogenetics and pharmacogenomics: Origin, status, and the hope for personalized medicine. Pharmacogenomics J 2006;6:162-5. [ Links ]
9. Pirmohamed M. Genetic factors in the predisposition to drug-induced hypersensitivity reactions. AAPS J 2006;8:E20-6. [ Links ]
10. Chen M, Xia B, Chen B, Guo Q, Li J, Ye M, et al. N –acetyltransferase 2 slow acetylator genotype associated with adverse effects of sulfasalazine in the treatment of inflammatory bowel disease. Can J Gastroenterol 2007;21:155-8. [ Links ]
11. Goodall R. Cholinesterase heterogeneity: Pharmacogenetic models and clinical implications. Current Anaesthesia and Critical Care 2004;15:29-35. [ Links ]
12. Reynolds KK, Valdes Jr R, Hartung BR, Linder MW. Individua lizing warfarin therapy. Personalized Medicine 2007;4:11-31. [ Links ]
13. Wang D, Curtis A, Papp AC, Koletar SL, Para MF. Polymorphism in glutamate cysteine ligase catalytic subunit (GCLC) is asso ciated with sulfamethoxazole -induced hypersensitivity in HIV/AIDS patients. BMC Med Genomics 2012;5:32. [ Links ]
14. Gueant JL, Gueant -Rodriguez RM, Gastin IA, Cornejo-Garcia JA, Viola M, Barbaud A, et al. Pharmacogenetic determinants of immediate and delayed reactions of drug hypersensitivity. Curr Pharm Des 2008;14:2770-7. [ Links ]
15. Apter AJ, Schelleman H, Walker A, Addya K, Rebbeck T. Clinical and genetic risk factors of self -reported penicillin allergy. J Allergy Clin Immunol 2008;122:152-8. [ Links ]
16. Guéant -Rodriguez RM, Romano A, Béri -Dexheimer M, Viola M, Gaeta F, Guéant JL. Gene -gene interactions of IL13 and IL4RA variants in immediate allergic reactions to betalactam antibiotics. Pharmacogenet Genomics 2006;16:713-9. [ Links ]
17. Huang CZ, Yang J, Qiao HL, Jia LJ. Polymorphisms and haplotype analysis of IL -4Ralpha Q576R and I75V in patients with penicillin allergy. Eur J Clin Pharmacol 2009;65:895-902. [ Links ]
18. Qiao HL, Yang J, Zhang YW. Relationships between specific serum IgE, cytokines and polymorphisms in the IL-4, IL-4Rα in patients with penicillins allergy. Allergy 2005;60:1053-9. [ Links ]
19. Qiao HL, Yang J, Zhang YW. Specific serum IgE levels and FcεRIβ genetic polymorphism in patients with penicillins allergy. Allergy 2004;59:1326-32. [ Links ]
20. Guglielmi L, Fontaine C, Gougat C, Avinens O, Eliaou JF, Guglielmi P, et al. IL -10 promoter and IL4 -Rα gene SNPs are associated with immediate β -lactam allergy in atopic women. Allergy 2006;61:921-7. [ Links ]
21. Kim SH, Lee JE, Kim SH, Jee YK, Kim YK, Park HS, et al. Allelic variants of CD40 and CD40L genes interact to promote antibiotic-induced cutaneous allergic reactions. Clin Exp Allergy 2009;39:1852-6. [ Links ]
22. Perry Jr HM, Tan EM, Carmody S, Sakamoto A. Relationship of acetyl transferase activity to antinuclear antibodies and toxic symptoms in hypertensive patients treated with hydralazine. J Lab Clin Med 1970;76:114-25. [ Links ]
23. Batchelor JR, Welsh KI, Mansilla Tinoco R, Dollery CT, Hughes GR, Bernstein R, et al. Hydralazine -induced systemic lupus erythematosus: Influence of HLA -DR and sex on susceptibility. Lancet 1980;1:1107-9. [ Links ]
24. Pirmohamed M, Park BK. Genetic susceptibility to adverse drug reactions. Trends Pharmacol Sci 2001;22:298-305. [ Links ]
25. Nolan D. HLA -B*5701 screening prior to abacavir prescription: Clinical and laboratory aspects. Crit Rev Clin Lab Sci 2009;46:153-65. [ Links ]
26. Chessman D, Kostenko L, Lethborg T, Purcell AW, Williamson NA, Chen Z, et al. Human leukocyte antigen class I-restricted activation of CD8+ T cells provides the immunogenetic basis of a systemic drug hypersensitivity. Immunity 2008;28:822-32. [ Links ]
27. Mallal S, Nolan D, Witt C, Masel G, Martin AM, Moore C, et al. Association between presence of HLA -B*5701, HLA-DR7, and HLA-DQ3 and hypersensitivity to HIV -1 reverse –transcriptase inhibitor abacavir. Lancet 2002;359:727 -32. [ Links ]
28. Hetherington S, Hughes AR, Mosteller M, Shortino D, Baker KL, Spreen W, et al. Genetic variations in HLA -B region and hypersensitivity reactions to abacavir. Lancet 2002;359:1121-2. [ Links ]
29. Martin AM, Nolan D, Gaudieri S, Almeida CA, Nolan R, James I, et al. Predisposition to abacavir hypersensitivity conferred by HLA -B*5701 and a haplotypic Hsp70 -Hom variant. Proc Natl Acad Sci USA 2004;101:4180-5. [ Links ]
30. Hughes AR, Mosteller M, Bansal AT, Davies K, Haneline SA, Lai EH, et al. Association of genetic variations in HLA -B region with hypersensitivity to abacavir in some, but not all, populations. Pharmacogenomics 2004;5:203-11. [ Links ]
31. Mallal S, Phillips E, Carosi G, Molina JM, Workman C, Tomazic J, et al. HLA -B*5701 screening for hypersensitivity to abacavir. N Engl J Med 2008;358:568-79. [ Links ]
32. Saag M, Balu R, Phillips E, Brachman P, Martorell C, Burman W, et al. High sensitivity of human leukocyte antigen-b*5701 as a marker for immunologically confirmed abacavir hypersensitivity in white and black patients. Clin Infec Dis 2008;46:1111-8. [ Links ]
33. Abacavir (marketed as Ziagen) and Abacavir -Containing Medications: FDA; 2008 [16 Novembro 2013]. Available from: http://www.fda.gov/Drugs/DrugSafety/PostmarketDrugSafetyInformationforPatientsandProviders/ucm123927.htm. [ Links ]
34. Kivexa: European Medicines Agency; 2009 [17 Novembro 2013]. Available from: http://www.ema.europa.eu/docs/en_GB/document_library/EPAR_-_Procedural_steps_taken_and_scientific_information_after_authorisation/human/000581/WC500043732.pdf. [ Links ]
35. Thompson MA, Aberg JA, Cahn P, Montaner JSG, Rizzardini G, Telenti A, et al. Antiretroviral treatment of adult HIV infection: 2010 recommendations of the International AIDS Society –USA panel. JAMA 2010;304:321-33. [ Links ]
36. Poggi H, Vera A, Lagos M, Solari S, Luis RP, Pérez CM. HLA -B*5701 frequency in Chilean HIV -infected patients and in general population. Braz J Infect Dis 2010;14:510-2. [ Links ]
37. Puthanakit T, Bunupuradah T, Kosalaraksa P, Vibol U, Hansudewechakul R, Ubolyam S, et al. Prevalence of human leukocyte antigen -B*5701 among HIV -infected children in Thailand and Cambodia: Implications for abacavir use. Pediatr Infect Dis J 2013;32:252-3. [ Links ]
38. Santos N, Pinheiro C, Carolino F, Faustino AS, Soares P, Sarmento A, et al. Prevalence of abacavir -associated hypersensiti vity syndrome and HLA -B*5701 allele in a Portuguese HIV population. Allergy 2013;68(Suppl.97):517. [ Links ]
39. To SWC, Chen JHK, Wong KH, Chan KCW, Tsang OTY, Yam WC. HLA -B*5701 genetic screening among HIV -1 infected patients in Hong Kong: Is this a practical approach in Han-Chinese? Int J STD AIDS 2013;24:50-2. [ Links ]
40. Munderi P, Snowden WB, Walker AS, Kityo C, Mosteller M, Kabuye G, et al. Distribution of HLA -B alleles in a Ugandan HIV-infected adult population: NORA pharmacogenetic substudy of DART. Trop Med Int Health 2011;16:200-4. [ Links ]
41. Schackman BR, Scott CA, Walensky RP, Losina E, Freedberg KA, Sax PE. The cost -effectiveness of HLA -B*5701 genetic screening to guide initial antiretroviral therapy for HIV. AIDS 2008;22:2025-33. [ Links ]
42. Wolf E, Blankenburg M, Bogner JR, Becker W, Gorriahn D, Mueller MC, et al. Cost impact of prospective HLA -B*5701 –screening prior to abacavir/lamivudine fixed dose combination use in Germany. Eur J Med Res 2010;15:145 -51. [ Links ]
43. Nieves Calatrava D, Calle -Martín ÓDL, Iribarren -Loyarte JA, Rivero -Román A, García -Bujalance L, Pérez -Escolano I, et al. Cost -effectiveness analysis of HLA -B*5701 typing in the prevention of hypersensitivity to abacavir in HIV patients in Spain. Enferm Infecc Microbiol Clin 2010;28:590-5. [ Links ]
44. Chung WH, Hung SI, Hong HS, Hsih MS, Yang LC, Ho HC, et al. Medical genetics: a marker for Stevens -Johnson syndrome. Nature 2004;428:486. [ Links ]
45. Lonjou C, Borot N, Sekula P, Ledger N, Thomas L, Halevy S, et al. A European study of HLA -B in Stevens-Johnson syndrome and toxic epidermal necrolysis related to five high -risk drugs. Pharmacogenet Genomics 2008;18:99 -107. [ Links ]
46. Man CB, Kwan P, Baum L, Yu E, Lau KM, Cheng AS, et al. Association between HLA -B*1502 allele and antiepileptic drug –induced cutaneous reactions in Han Chinese. Epilepsia 2007;48:1015-8. [ Links ]
47. Locharernkul C, Loplumlert J, Limotai C, Korkij W, Desudchit T, Tongkobpetch S, et al. Carbamazepine and phenytoin induced Stevens -Johnson syndrome is associated with HLA -B*1502 allele in Thai population. Epilepsia 2008;49:2087 -91. [ Links ]
48. Mehta TY, Prajapati LM, Mittal B, Joshi CG, Sheth JJ, Patel DB, et al. Association of HLA -B*1502 allele and carbamazepine-induced Stevens -Johnson syndrome among Indians. Indian J Dermatol Venereol Leprol 2009;75:579-82. [ Links ]
49. Chang CC, Too CL, Murad S, Hussein SH. Association of HLA-B*1502 allele with carbamazepine -induced toxic epidermal necrolysis and Stevens -Johnson syndrome in the multi –ethnic Malaysian population. Int J Dermatol 2011;50:221 -4. [ Links ]
50. Information for healthcare professionals: Dangerous or even fatal skin reactions – Carbamazepine: FDA; 2007 [17 Novembro 2013]. Available from: http://www.fda.gov/Drugs/DrugSafety/PostmarketDrugSafetyInformationforPatientsandProviders/ucm124718.htm [ Links ]
51. Dong D, Sung C, Finkelstein EA. Cost -effectiveness of HLA-B*1502 genotyping in adult patients with newly diagnosed epilepsy in Singapore. Neurology 2012;79:1259-67. [ Links ]
52. Alfirevic A, Jorgensen AL, Williamson PR, Chadwick DW, Park BK, Pirmohamed M. HLA -B locus in Caucasian patients with carbamazepine hypersensitivity. Pharmacogenomics 2006;7:813-8. [ Links ]
53. Kim SH, Lee KW, Song WJ, Kim SH, Jee YK, Lee SM, et al. Carbamazepine -induced severe cutaneous adverse reactions and HLA genotypes in Koreans. Epilepsy Res 2011;97:190-7. [ Links ]
54. Kaniwa N, Saito Y, Aihara M, Matsunaga K, Tohkin M, Kurose K, et al. HLA-B locus in Japanese patients with anti -epileptics and allopurinol-related Stevens Johnson syndrome and toxic epidermal necrolysis. Pharmacogenomics 2008;9:1617-22. [ Links ]
55. Ozeki T, Mushiroda T, Yowang A, Takahashi A, Kubo M, Shirakata Y, et al. Genome -wide association study identifies HLA-A*3101 allele as a genetic risk factor for carbamazepine-induced cutaneous adverse drug reactions in Japanese population. Hum Mol Genet 2011;20:1034-41. [ Links ]
56. Hung SI, Chung WH, Jee SH, Chen WC, Chang YT, Lee WR, et al. Genetic susceptibility to carbamazepine-induced cutaneous adverse drug reactions. Pharmacogenet Genomics 2006;16:297-306. [ Links ]
57. Alfirevic A, Mills T, Harrington P, Pinel T, Sherwood J, Jawaid A, et al. Serious carbamazepine -induced hypersensitivity reactions associated with the HSP70 gene cluster. Pharmacogenet Geno mics 2006;16:287-96. [ Links ]
58. Hung SL, Chung WH, Liou LB, Chu CC, Lin M, Huang HP, et al. HLA -B*5801 allele as a genetic marker for severe cutaneous adverse reactions caused by allopurinol. Proc Natl Acad Sci USA 2005;102:4134-9. [ Links ]
59. Tassaneeyakul W, Jantararoungtong T, Chen P, Lin PY, Tiamkao S, Khunarkornsiri U, et al. Strong association between HLA-B*5801 and allopurinol -induced Stevens -Johnson syndrome and toxic epidermal necrolysis in a Thai population. Pharmacogenet Genomics 2009;19:704-9. [ Links ]
60. Kang HR, Jee YK, Kim YS, Lee CH, Jung JW, Kim SH, et al. Positive and negative associations of HLA class i alleles with allopurinol-induced SCARs in Koreans. Pharmacogenet Genomics 2011;21:303-7. [ Links ]
61. Gonçalo M, Coutinho I, Teixeira V, Gameiro AR, Brites MM, Nunes R, et al. HLA -B*58:01 is a risk factor for allopurinol-induced DRESS and Stevens -Johnson syndrome/toxic epidermal necrolysis in a Portuguese population. Br J Dermatol 2013;169:660-5. [ Links ]
62. Kim SH, Ye YM, Palikhe NS, Kim JE, Park HS. Genetic and ethnic risk factors associated with drug hypersensitivity. Curr Opin Allergy Clin Immunol 2010;10:280-90. [ Links ]
63. Hershfield MS, Callaghan JT, Tassaneeyakul W, Mushiroda T, Thorn CF, Klein TE, et al. Clinical Pharmacogenetics Implementation Consortium guidelines for human leukocyte antigen -B genotype and allopurinol dosing. Clin Pharmacol Ther 2013;93:153-8. [ Links ]
64. Kindt TJ. Kuby Immunology. 6th ed. New York: W.H. Freeman; 2007. [ Links ]
65. Yun J, Adam J, Yerly D, Pichler WJ. Human leukocyte antigens (HLA) associated drug hypersensitivity: Consequences of drug binding to HLA. Allergy 2012;67:1338-46. [ Links ]
66. Adkinson Jr NF, Essayan D, Gruchalla R, Haggerty H, Kawabata T, Sandler JD, et al. Task force report: Future research needs for the prevention and management of immune -mediated drug hypersensitivity reactions. J Allergy Clin Immunol 2002;109:S461-78. [ Links ]
67. Illing PT, Vivian JP, Purcell AW, Rossjohn J, McCluskey J. Human leukocyte antigen -associated drug hypersensitivity. Curr Opin Immunol 2013;25:81-9. [ Links ]
68. Jenneck C, Juergens U, Buecheler M, Novak N. Pathogenesis, diagnosis, and treatment of aspirin intolerance. Ann Allergy Asthma Immunol 2007;99:13-21. [ Links ]
69. Ayuso P, Blanca -López N, Doña I, Torres MJ, Guéant –Rodríguez RM, Canto G, et al. Advanced phenotyping in hypersensitivity drug reactions to NSAIDs. Clin Exp Allergy 2013;43:1097-109. [ Links ]
70. Choi JH, Lee KW, Oh HB, Lee KJ, Suh YJ, Park CS, et al. HLA association in aspirin -intolerant asthma: DPB1*0301 as a strong marker in a Korean population. J Allergy Clin Immunology 2004;113:562-4. [ Links ]
71. Park SM, Park JS, Park HS, Park CS. Unraveling the genetic basis of aspirin hypersensitivity in asthma beyond arachidonate pathways. Allergy Asthma Immunology Res 2013;5:258-76. [ Links ]
72. Shrestha Palikhe N, Kim SH, Jin HJ, Hwang EK, Nam YH, Park HS. Genetic mechanisms in aspirin -exacerbated respiratory disease. J Allergy 2012;2012:794890. [ Links ]
73. Kim SH, Choi JH, Lee KW, Kim SH, Shin ES, Oh HB, et al. The human leucocyte antigen -DRB1*1302 -DQB1*0609 -DPB1*0201 haplotype may be a strong genetic marker for aspirin –induced urticaria. Clin Exp Allergy 2005;35:339-44. [ Links ]
74. Kowalski ML, Woszczek G, Bienkiewicz B, Mis M. Association of pyrazolone drug hypersensitivity with HLA -DQ and DR antigens. Clin Exp Allergy 1998;28:1153-8. [ Links ]
75. Palikhe NS, Kim SH, Cho BY, Ye YM, Choi GS, Park HS. Genetic variability in CRTH2 polymorphism increases eotaxin-2 levels in patients with aspirin exacerbated respiratory disease. Allergy 2010;65:338-46. [ Links ]
76. Lee JS, Kim JH, Bae JS, Kim JY, Park TJ, Pasaje CF, et al. Association of CACNG6 polymorphisms with aspirin -intolerance asthmatics in a Korean population. BMC Med Genet 2010;11:138. [ Links ]
77. Sánchez -Borges M, Acevedo N, Vergara C, Jiménez S, Zabner-Oziel P, Monzón A, et al. The A-444C polymorphism in the leukotriene C4 synthase gene is associated with aspirin–induced urticaria. J Investig Allergol Clin Immunol 2009;19:375-82. [ Links ]
78. Cornejo -Garcia JA, Jagemann LR, Blanca -Lopez N, Dona I, Flores C, Gueant -Rodriguez RM, et al. Genetic variants of the arachidonic acid pathway in non-steroidal anti-inflammatory drug-induced acute urticaria. Clin Exp Allergy 2012;42:1772-81. [ Links ]
79. Palikhe NS, Kim SH, Lee HY, Kim JH, Ye YM, Park HS. Association of thromboxane A2 receptor (TBXA2R) gene polymorphism in patients with aspirin -intolerant acute urticaria. Clin Exp Allergy 2011;41:179-85. [ Links ]
80. Palikhe NS, Kim SH, Cho BY, Ye YM, Hur GY, Park HS. Association of three sets of high -affinity IgE receptor (FcepsilonR1) polymorphisms with aspirin -intolerant asthma. Respir Med 2008;102:1132-9. [ Links ]
81. Bae JS, Kim SH, Ye YM, Yoon HJ, Suh CH, Nahm DH, et al. Significant association of FcεRIα promoter polymorphisms with aspirin-intolerant chronic urticaria. J Allergy Clin Immunol 2007;119:449-56. [ Links ]
82. Kim SH, Kang YM, Kim SH, Cho BY, Ye YM, Hur GY, et al. Histamine N -methyltransferase 939A>G polymorphism affects mRNA stability in patients with acetylsalicylic acid –intolerant chronic urticaria. Allergy 2009;64:213-21. [ Links ]
83. Bousquet PJ, Demoly P, Romano A, Aberer W, Bircher A, Blanca M, et al. Pharmacovigilance of drug allergy and hypersensitivity using the ENDA -DAHD database and the GA2LEN platform. The Galenda project. Allergy 2009;64:194-203. [ Links ]
84. ODonohue J, Oien KA, Donaldson P, Underhill J, Clare M, Mac-Sween RNM, et al. Co -amoxiclav jaundice: Clinical and histological features and HLA class II association. Gut 2000;47:717-20. [ Links ]
85. Vitezica ZG, Milpied B, Lonjou C, Borot N, Ledger TN, Lefebvre A, et al. HLA -DRB1*01 associated with cutaneous hypersensiti vity induced by nevirapine and efavirenz. AIDS 2008;22:540-1. [ Links ]
86. Daly AK, Donaldson PT, Bhatnagar P, Shen Y, PeEr I, Floratos A, et al. HLA -B5701 genotype is a major determinant of drug –induced liver injury due to flucloxacillin. Nat Genet 2009;41:816-9. [ Links ]
87. Chantarangsu S, Mushiroda T, Mahasirimongkol S, Kiertiburanakul S, Sungkanuparph S, Manosuthi W, et al. HLA -B* 3505 allele is a strong predictor for nevirapine-induced skin adverse drug reactions in HIV -infected Thai patients. Pharmacogenet Geno mics. 2009;19:139-46. [ Links ]
88. Littera R, Carcassi C, Masala A, Piano P, Serra P, Ortu F, et al. HLA -dependent hypersensitivity to nevirapine in Sardinian HIV patients. AIDS 2006;20:1621-6. [ Links ]
89. Shankarkumar U, Shah KN, Ghosh K. Letter: HLA B*1502 allele association with oxcarbamazepine -induced skin reactions in epilepsy patient from India. Epilepsia 2009;50:1837-8. [ Links ]
Natacha Santos
Serviço de Imunoalergologia, Centro Hospitalar São João
Alameda Professor Hernâni Monteiro
4202 -451 Porto
E-mail: natachalsantos@gmail.com
Financiamento: Nenhum.
Declaração de conflito de interesses: Nenhum.
Data de recepção/ Received in: 10/12/2013
Data de aceitação / Accepted for publication in: 26/12/2013