Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Portuguesa de Ortopedia e Traumatologia
versão impressa ISSN 1646-2122
Rev. Port. Ortop. Traum. vol.21 no.4 Lisboa dez. 2013
INVESTIGAÇÃO
Isolamento e caracterização das células da notocorda humana. Implicações para o desenvolvimento de terapias celulares para a doença degenerativa discal
Ricardo Rodrigues-PintoI, II; António OliveiraI; Stephen M. RichardsonII; Judith A. HoylandII
I. Spine Research Group. Centre for Tissue Injury and Repair. Institute of Inflammation and Repair. Faculty of Medical and Human Sciences. Universidade de Manchester. Reino Unido.
II. Serviço de Ortopedia. Centro Hospitalar do Porto. Hospital Santo António. Porto. Portugal.
RESUMO
Introdução: Para o desenvolvimento de terapêuticas celulares para o tratamento da degeneração do disco intervertebral é necessário o conhecimento detalhado das células que o constituem e das vias moleculares que levam à sua diferenciação. O núcleo pulposo (NP) embrionário, fetal e dos primeiros anos de vida é formado por células grandes e vacuoladas da notocorda; no entanto, com a maturidade esquelética, as células da notocorda desaparecem e são substituídas por células menores, semelhantes a condrócitos. Estudos animais e in vitro demonstraram que as células da notocorda exercem efeitos anabólicos e protetores sobre o disco intervertebral, pelo que o seu desaparecimento do NP humano tem sido implicado na patogénese da degeneração discal. Deste modo, o estudo destas células e dos seus mecanismos de regulação poderá identificar fatores envolvidos na homeostasia do disco intervertebral e ajudar no desenvolvimento de terapêuticas celulares para regenerar ou substituir as células do disco intervertebral degenerado. No entanto, o estudo de células da notocorda humana tem sido dificultado por restrições éticas, logísticas e técnicas, o que constitui uma importante limitação para o avanço científico nesta área. Este estudo foi realizado com o objetivo de isolar células da notocorda (precursoras do NP) das do esclerótomo (precursoras do anel fibroso (AF) e das vértebras) a partir de embriões e fetos humanos e caracterizar o seu fenótipo e os mecanismos ou moléculas reguladores da sua função no disco intervertebral.
Métodos: Embriões e fetos humanos (3.5 a 12 semanas pósconceção) foram dissecados e caracterizados morfologicamente para confirmar a presença de células da notocorda na coluna vertebral em desenvolvimento. Uma vez que nesta altura do desenvolvimento humano não existe uma demarcação óbvia dos limites entre o NP, o AF e o corpo vertebral, foi desenvolvida uma estratégia baseada na identificação de um marcador celular específico para as células da notocorda e na sua utilização para as separar das células do esclerótomo. Para identificar esse marcador realizou-se uma pesquisa da literatura; seguidamente, os potenciais marcadores foram testados por imunohistoquímica em secções embrionárias e fetais contendo células da notocorda. Após identificação do marcador a usar, e uma vez que esse tinha localização intracelular, foi desenvolvida uma metodologia que permitisse extrair RNA a partir de um número limitado de células fixadas, permeabilizadas, marcadas com um anticorpo fluorescente e separadas por citometria de fluxo. O método desenvolvido foi então utilizado para separar e extrair RNA a partir de células da notocorda e esclerótomo fetais. A precisão da separação foi confirmada pela quantificação por reação em cadeia da polimerase em tempo real (qRT-PCR) da expressão de genes da notocorda nos dois tipos celulares separados. Posteriormente, microarrays foram utilizados para identificar o fenótipo e o software interactive pathway analysis (IPA®) foi utilizado para identificar moléculas e fatores reguladores da função das células da notocorda.
Resultados: A coluna vertebral fetal humana foi validada como uma fonte adequada para obtenção de células da notocorda. A pesquisa da literatura identificou 16 potenciais marcadores específicos destas células e a validação de um grupo deles identificou as moléculas KRT8, KRT18 e KRT19 como sendo específicas para as células da notocorda da coluna vertebral fetal humana. Verificou-se que uma metodologia consistindo na fixação e permeabilização de células com 95% etanol/ 5% ácido acético, marcação com um anticorpo fluorescente anti-KRT18 e separação por citometria de fluxo permitia obter RNA de elevada qualidade. Esse método foi utilizado para separar células extraídas enzimaticamente da coluna fetal humana e separar as células da notocorda (precursoras do NP e KRT18-positivas) das células do esclerótomo (precursoras do AF e das vértebras e KRT18-negativas). A maior expressão diferencial dos genes da notocorda KRT18, KRT19, brachyury, Galectin 3, CTGF e FOXA2 pelas células KRT18-positivas confirmou a precisão da separação. A análise dos dois tipos celulares por microarrays identificou 782 genes regulados positivamente e 678 regulados negativamente pelas células da notocorda. A análise IPA identificou 30 moléculas reguladoras dos genes expressos pelas células da notocorda. Destas, o hepatocyte growth fator (HGF) foi identificado como o principal fator de crescimento a montante das células da notocorda ativando genes importantes para a sua função no disco intervertebral.
Discussão: Este estudo isolou pela primeira vez células da notocorda humana. Para o realizar foi desenvolvida uma metodologia complexa, cujas aplicações se estendem para lá desta área de investigação. A análise por microarrays identificou marcadores fenotípicos das células da notocorda, alguns dos quais potencialmente responsáveis pelos seus efeitos protetores no disco intervertebral. O fator de crescimento HGF foi identificado como um regulador destas células, podendo ser responsável por algumas das funções que lhes são atribuídas.
Palavras chave: Disco intervertebral, núcleo pulposo, degeneração discal, células da notocorda, fenótipo, microarrays, regeneração, terapêuticas celulares.
ABSTRACT
Introduction: For the development of cell-based therapies targeting intervertebral disc (IVD) degeneration a thorough understanding of its cells and the molecular pathways leading to their differentiation is required. The embryonic, foetal and juvenile nucleus pulposus (NP) is populated by large vacuolated notochordal cells that with skeletal maturity become replaced by smaller chondrocyte-like cells. Since animal and in vitro studies have shown that notochordal cells display protective and anabolic roles in the IVD, their loss in humans has often been suggested to initiate the degenerative process. As such, a detailed understanding of these cells and their regulatory pathways may help to identify factors involved in IVD homeostasis and the development of novel cell-based therapies targeting disc degeneration. The study of human notochordal cells has however been hindered by ethical, logistical and technical restrictions in obtaining suitable samples, which constitutes a major limitation to the field. This study was conducted with the objective of isolating notochordal cells (precursors of the NP) from their adjacent sclerotomal tissues (precursors of the annulus fibrosus (AF) and vertebrae) from human embryonic and foetal spines and to identify their phenotype and potential factors involved in their function in the IVD.
Methods: Human embryonic and foetal spines (3.5-12 weeks post-conception) were dissected and characterised morphologically to confirm their suitability as sources of notochordal cells. Then, and since there was no visible demarcation between the NP, AF and vertebral regions, a strategy involving the identification of a notochord-specific marker to label and sort notochordal cells was devised. To identify a notochordspecific marker, a literature search was performed followed by the validation of identified putative markers in the human foetal spine using immunohistochemistry. Subsequently, and as the identified marker had an intracellular location, a novel methodology was developed to allow for the extraction of high quality RNA from limited numbers of fixed, permeabilised, labelled with a fluorescent antibody and sorted cells. Using the optimised methodology, RNA was extracted from notochordal and sclerotomal cells. Separation accuracy was validated using real time quantitative polymerase chain reaction (qRT-PCR) for known notochordal markers. Microarrays were then used to identify differentially expressed genes between notochordal and sclerotomal cells and interactive pathway analysis (IPA) was used to identify notochordal cell regulators.
Results: The human foetal spine was validated as a suitable source of notochordal cells. The literature search identified 16 putative markers and the validation of a panel of these identified KRT8, KRT18 and KRT19 as human notochord-specific markers. A methodology involving cell fixation and permeabilisation using 95% ethanol/ 5% acetic acid, followed by incubation with a directly conjugated KRT18 antibody and separation by fluorescence activated cell sorting was found to have no detrimental effect on the RNA quality. This method was therefore used to separate enzymatically obtained human foetal notochordal cells (NP precursors and KRT18-positive) from sclerotomal cells (AF and vertebrae precursors and KRT18-negative). qRT-PCR analysis validated the cell separation methodology by confirming that notochordal cells had higher differential expression of the notochordal markers KRT18, KRT19, brachyury, Galectin 3, CTGF and FOXA2. Array analysis identified 782 upregulated and 678 downregulated genes. IPA analysis identified 30 upstream regulators of notochordal cell genes. Of these, hepatocyte growth factor (HGF) was the top growth factor and was predicted to be activating a number of relevant notochordal genes.
Discussion: This study isolated, for the first time, human notochordal cells. To do so, a complex labelling methodology was devised that is applicable to different areas of research. Microarray analysis identified a list of notochordal cell markers, some of which may be important in notochordal cell function within the IVD. Importantly, HGF was identified as an upstream regulator of notochordal cells that may be involved in some of the biological functions attributed to them.
Key words: Intervertebral disc, nucleus pulposus, disc degeneration, notochordal cells, phenotype, microarrays, regeneration, cellular therapies.
INTRODUÇÃO
A dor lombar é um sintoma praticamente universal, apresentando prevalências pontual, mensal e global de 11.9%[1], 23.2% e 84%[2], respetivamente. Apesar de fatores como carga, hereditariedade, profissão, diabetes, obesidade, tabagismo e consumo de álcool estarem implicados na sua patogénese, estudos observacionais demonstram que cerca de 40% dos casos de lombalgia têm associada degeneração do disco intervertebral (diagnosticada por ressonância magnética)[3, 4], tendo já sido estabelecida uma relação de causalidade entre os dois fenómenos[5, 6].
O disco intervertebral é uma estrutura complexa que permite flexibilidade à coluna vertebral e suporta e distribui o peso que é aplicado através desta. É formado perifericamente pelo anel fibroso (AF), uma estrutura lamelar formada sobretudo por colagénio tipo I que rodeia o núcleo pulposo (NP), uma estrutura gelatinosa, hidratada e rica em proteoglicanos. A separar o disco intervertebral das vértebras adjacentes encontram-se placas terminais cartilagíneas, que permitem o transporte passivo de nutrientes da vértebra para o disco e de produtos de degradação do disco para a vértebra[7]. Apesar das densidades celulares no NP e no AF serem extremamente baixas[8] as suas células assumem um papel fundamental na manutenção da integridade do disco intervertebral, sendo responsáveis pela síntese e degradação da matriz extracelular destas estruturas[8]. Isto assume particular importância no NP, que é a primeira estrutura a ser afetada durante a degeneração discal e, consequentemente, aquela cuja integridade funcional é mais importante para a manutenção da função normal do disco intervertebral[9].
A degeneração discal inicia-se assim, por um desequilíbrio entre a síntese e degradação da matriz extracelular do NP mediada pelas suas células e que se caracteriza por uma redução do conteúdo de proteoglicanos e uma substituição gradual de colagénio tipo II (o principal colagénio do NP) pelo mais fibroso colagénio tipo I. Estas alterações celulares e da matriz extracelular levam à desidratação do NP, que se torna incapaz de distribuir equitativamente as forças compressivas provenientes dos corpos vertebrais, e que são consequentemente transmitidas de forma não uniforme para o AF gerando áreas de pressão aumentada e micro-trauma[10]. Estas alterações na normal distribuição de cargas levam ao aparecimento de fissuras e roturas no AF, através das quais o NP hernia para o canal vertebral e neovasos e nervos crescem para o interior do disco; estes vasos e nervos transportam estímulos nocicetivos que contribuem para a dor lombar[6]. Em última instância, a altura do disco intervertebral diminui. Esta cadeia de eventos envolve também as estruturas anatómicas vizinhas, uma vez que a perda de altura do disco e da sua capacidade de suportar cargas levam ao aumento da carga sobre as facetas articulares intervertebrais e o ligamento amarelo, que hipertrofiam, e sobre a musculatura paravertebral, originando desequilíbrios musculares; o conjunto destes fatores é responsável pela dor lombar axial. Por outro lado, o abaulamento, herniação e perda de altura discais e a hipertrofia de facetas e do ligamento amarelo reduzem o diâmetro do canal vertebral e dos buracos vertebrais, levando à compressão medular e/ou das raízes nervosas[11, 12].
Quando as medidas de tratamento conservador para as patologias associadas à degeneração discal (herniação discal, estenose canalar com ou sem espondilolistese e escoliose degenerativa[11, 12]) falham, o tratamento cirúrgico consiste frequentemente na exérese do disco e consequente fusão do segmento vertebral ou a sua substituição protésica. Apesar destas terapêuticas serem relativamente eficazes no alívio da dor e terem resultados clínicos geralmente favoráveis, estão também associadas a complicações importantes, como a degeneração do disco adjacente ou complicações relacionadas com o implante, tais como migração, infeção ou falência[13, 14]. Além disso, estes tratamentos estão direcionados para o alívio sintomático mas não resolvem a patologia subjacente.
As terapêuticas celulares constituem alternativas ou complementos aos tratamentos cirúrgicos disponíveis atualmente. Em Ortopedia, estas estratégias foram já aplicadas com bons resultados na regeneração tendinosa[15], meniscal[16], ligamentar[17], cartilagínea[18] e óssea[19]. A terapia celular baseia-se em dois métodos fundamentais:
i) ex vivo, em que o tecido é completamente gerado in vitro e só então transplantado para o doente;
ii) in vivo, em que células isoladamente ou em combinação com estruturas tridimensionais porosas (scaffolds) são implantadas sem terem sido cultivadas previamente, maturando e diferenciando-se no corpo do doente[20]. Para o sucesso de qualquer dos métodos é crucial que as células implantadas sejam capazes de substituir e/ ou estimular as células nativas a produzirem uma matriz extracelular saudável e hidratada, capaz de realizar as suas funções.
As células estaminais mesenquimatosas (MSC) são células estaminais do adulto que se encontram em múltiplos tecidos (medula óssea, tecido adiposo, cordão umbilical, músculo, derme, periósseo, membrana e líquido sinoviais e cartilagem[21-23]) e que podem diferenciar-se em células de diversos tecidos como cartilagem [21, 24, 25], osso[25-27], tecido adiposo[21, 28], ligamento[23, 29] e músculo[21]. Estudos na área do disco intervertebral mostram que as MSC têm potencial para regenerar o disco intervertebral[30-35]. No entanto, para que uma célula progenitora seja diferenciada e substitua e estimule a população celular do NP, é essencial que o fenótipo da célula do NP seja conhecido. Só este conhecimento permitirá que a célula implantada execute as suas funções, produzindo e mantendo uma matriz extracelular hidratada.
Recentemente, o fenótipo das células dos NP adulto foi elucidado usando microarrays [36, 37]. As células do NP são, no entanto, complexas na sua morfologia, fenótipo e origem embrionária. Enquanto que o NP juvenil é populado por células grandes e vacuoladas derivadas da notocorda fetal e embrionária, com a maturidade esquelética estas células da notocorda “desaparecem”, sendo substituídas por células mais pequenas, semelhantes a condrócitos, que têm sido chamadas de células do NP adulto. Este fenómeno é particularmente importante uma vez que a “perda” de células da notocorda tem sido implicada na patogénese da degeneração discal, tal como é sugerido pela análise post-mortem de discos intervertebrais realizada por Walmsley em que se verificou que o momento do desaparecimento destas células se correlaciona com o aparecimento dos primeiros sinais histológicos de degeneração[38]. Esta hipótese é ainda suportada pelo facto de certas raças de cães (denominadas condrodistróficas), tal como o homem, perderem as células da notocorda e desenvolverem espontaneamente degeneração discal, enquanto que outras raças (não-condrodistróficas), que mantêm essas células ao longo da vida, são resistentes à degeneração discal[39-42]. In vitro, existe evidência de que as células da notocorda têm um papel importante na manutenção da homeostasia do disco intervertebral: estas produzem mais proteoglicanos do que as células do NP adulto e, portanto, uma matriz extracelular mais hidratada[43]; quando em co-cultura as células da notocorda estimulam as células do NP adulto a produzirem uma matriz extracelular mais hidratada[39, 44] e evitam a morte das células do NP adulto induzida pela interleucina-1 (Il-1)[45].
Se, tal como a evidência existente sugere, as células da notocorda ou fatores por elas sintetizados são os ideais para manter uma matriz do disco intervertebral hidratada e funcionante, o conhecimento do fenótipo destas células e das moléculas e mecanismos que regulam o seu desenvolvimento, permitirá identificar fatores importantes na manutenção da homeostasia do disco intervertebral e no desenvolvimento de novas terapêuticas regenerativas/celulares. No entanto, e como as células da notocorda apenas estão presentes no disco intervertebral durante os períodos embrionário e fetal e nos primeiros anos após o nascimento, a investigação destas células tem sido limitada por restrições éticas, logísticas e técnicas e, até à data, não existe qualquer estudo caracterizando as células da notocorda humana.
A notocorda humana origina-se após a 3ª semana pós-conceção (SPC). É formada por células grandes e vacuoladas e ocupa uma posição central ao longo do eixo crânio-caudal do embrião, encontrandose anterior ao tubo neural e com uma coluna de sómitos em cada lado. Após a 4ª SPC a notocorda induz a migração central das células dos sómitos (então denominadas células do esclerótomo), para circundarem e envolverem a notocorda e o tubo neural[46]. Durante as 5ª e 6ª SPC, as células do esclerótomo adotam uma organização segmentar, com segmentos de elevada densidade celular intercalando com segmentos de menor densidade; centralmente, mantém-se presente uma coluna contínua de células da notocorda, que suporta o embrião. Após a 7ª SPC as células do esclerótomo nas regiões de menor densidade celular começam a comprimir a região central, “empurrando” as células da notocorda para os segmentos adjacentes. Nestes, as células do esclerótomo vão condensar-se na periferia, adotando uma distribuição lamelar, acomodando no seu centro as células da notocorda provenientes dos segmentos adjacentes. Nesta fase, as estruturas vertebrais assumem já as características morfológicas da coluna do adulto: as células centrais da notocorda correspondem ao NP, as células do esclerótomo localizadas à periferia destas constituirão o AF e as células do esclerótomo nos segmentos adjacentes começam o processo de hipertrofia que as levará a formação do corpo vertebral[47, 48].
A hipótese para este estudo foi que a coluna embrionária e fetal humana contém células da notocorda e que a identificação do fenótipo destas células e dos fatores por elas produzidos são importantes para o desenvolvimento de terapêuticas celulares para a regeneração do disco intervertebral. Para testar esta hipótese, células da notocorda humana foram isoladas da coluna vertebral fetal e microarrays foram utilizados para caracterizar o seu fenótipo e identificar fatores responsáveis pela sua função no disco intervertebral.
MATERIAL E MÉTODOS
Aquisição de amostras e cultura celular
Embriões (3ª-8ª SPC) e fetos (8ª-12ª SPC) humanos foram adquiridos através de uma colaboração com o grupo do Professor Neil Hanley (Institute of Human Development, Faculty of Medical and Human Sciences, The University of Manchester, Reino Unido) após consentimento autorizado por parte de mulheres a realizarem interrupções voluntárias da gravidez. Aprovação para a realização do projeto foi obtida junto do comité de ética local (Manchester Royal Infirmary, Ref. No: 08/H1010/28 Early Pregnancy Tissue Collection). Os embriões foram estadiados por esteromicroscopia de acordo com a classificação de Carnegie[49], sendo esta posteriormente convertida em SPC. O comprimento das mãos e pés fetais foram usados para dar uma estimativa da idade destes em SPC.
As células MCF-7 (Michigan Cancer Foundation) são uma linha celular de células de carcinoma mamário. Um recipiente contendo 4x106 células MCF-7 foi adquirido através da European Collection of Cell Cultures. Estas células foram cultivadas a 37ºC e 5% CO2 em EMEM contendo 10% de soro fetal bovino, 1% de glutamina, 1% de aminoácidos não essenciais, 100U/ml de penicilina, 100mg/ml de estreptomicina e 250ng de anfotericina.
Dissecção de colunas vertebrais embrionárias e fetais humanas
Devido à pequena dimensão dos embriões e fetos e à inexistência de uma demarcação clara entre os limites do NP, AF e corpo vertebral em desenvolvimento, a dissecção foi realizada para isolar as estruturas precursoras da coluna vertebral (vértebras e discos intervertebrais) desde a região cervical à lombar. Deste modo, colunas vertebrais juntamente com os seus tecidos adjacentes (espinhal medula, costelas e ligamentos) foram colhidos do embrião/feto e transferidos para uma placa de Petri contendo solução tampão salina de fosfato (PBS) Posteriormente, as costelas, nas suas articulações costovertebrais, e a medula espinhal primeiro, e os ligamentos longitudinais anterior e posterior depois foram cuidadosamente dissecados da coluna vertebral e descartados. Por último, as colunas vertebrais dissecadas contendo apenas os discos intervertebrais e as vértebras foram lavadas em PBS (Figura 1).
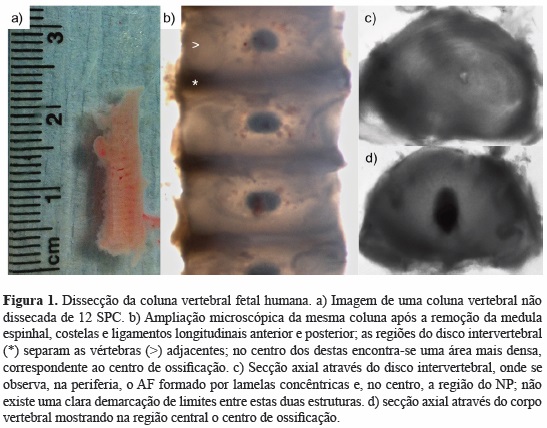
Caracterização histológica da coluna vertebral embrionária e fetal
As colunas vertebrais dissecadas foram imediatamente fixadas em 4% paraformaldeído/PBS, descalcificadas em 20% EDTA, re-hidratadas em água, processadas e embebidas em parafina. Secções sagitais com 5µm de espessura ao longo de toda a coluna foram obtidas e montadas em lâminas para visualização. A coloração de hematoxilina e eosina (H&E) foi usada para caracterização morfológica da coluna vertebral e identificação das regiões contendo células da notocorda. As lâminas coradas foram visualizadas por microscopia de luz.
Pesquisa da literatura
Devido à ausência de uma demarcação clara entre os limites das diferentes estruturas vertebrais, foi realizada uma pesquisa literária para identificar um marcador específico das células da notocorda que pudesse ser utilizado para separá-las das células do esclerótomo adjacentes. As bases de dados da PubMed e Embase foram pesquisadas usando a seguinte combinação de termos: (“nucleus pulposus” OR “notochordal cells” OR “notochord”) AND (“marker” OR “gene” OR “protein” OR “phenotype”). Para complementar a pesquisa, referências relevantes dos artigos selecionados e de revisões da literatura foram também pesquisadas. Foram incluídos todos os estudos publicados na língua inglesa, portuguesa, espanhola e francesa, realizados em humanos ou animais. Os artigos selecionados foram pesquisados para identificar potenciais marcadores (genes ou proteínas) com expressão em células da notocorda (de animais que as retêm), em células do NP humano (que apesar de não possuir células da notocorda é derivado delas[50]) ou em células da notocorda de embriões ou fetos (independentemente da espécie a que pertencem).
Identificação imunohistoquímica de um marcador específico para as células da notocorda humana
Para identificar um marcador específico para as células da notocorda humana, um conjunto de marcadores identificados na pesquisa literária foi testado por imunohistoquímica em secções da coluna embrionária e fetal. A coloração foi realizada segundo o método de Avidina-Biotina-Peroxidase para as proteínas brain acid soluble protein 1 (BASP1), galectin-3 (Gal-3), CD55, connective tissue growth fator (CTGF), cytokeratin (KRT) 8, 18 e 19 e n-cadherin (NCAD). Imunoglobulinas (Ig) de controlo foram usadas como controlos negativos e secções de tecidos que expressam o anticorpo de interesse foram usadas como controlos positivos. Vários protocolos de recuperação antigénica e várias concentrações foram testados para determinar o método ideal para cada anticorpo. O quadro I detalha os anticorpos (primários e secundários) usados, bem como como as concentrações e protocolos de recuperação antigénica otimizados para cada anticorpo. Após a otimização destes, cada anticorpo foi testado numa coorte de secções de coluna vertebral embrionárias e fetais (3.5 - 12 SPC) contendo regiões da notocorda, para avaliar a especificidade de cada um dos anticorpos. As lâminas coradas foram visualizadas por microscopia de luz.
Extração de células da coluna vertebral embrionária/fetal
Imediatamente após a dissecção, as colunas vertebrais embrionárias e fetais foram cuidadosamente trituradas com um bisturi e digeridas durante 16 horas em a-MEM (Sigma-Aldrich®, M4526) contendo 100U/ml de penicilina, 100mg/mL de estreptomicina, 250ng de anfotericina, 2.5mg/mL de colagenase tipo II (Life Technologies®, 17101) e 1 mg/mL de hialuronidase (Sigma-Aldrich®, H3506). Após a digestão, as células foram ressuspendidas em Cell Dissociation Solution Non-enzymatic (Sigma-Aldrich®, C5789) a 37ºC por uma hora para dissociar os agregados de células da notocorda[51]. Após dissociação as células frescas isoladas da coluna vertebral dissecada foram lavadas em PBS e usadas para separação entre células da notocorda e do esclerótomo.
Desenvolvimento de uma metodologia para obtenção de RNA a partir de células da notocorda e do esclerótomo humanos
Dos marcadores testados por imunohistoquímica, a KRT18 foi identificada como sendo o marcador específico mais adequado para marcar e separar as células da notocorda (KRT18-positivas) das células do esclerótomo (KRT18-negativas) isoladas da coluna vertebral embrionária/ fetal. Uma vez que a sua localização é intracelular, para que as células sejam marcadas com um anticorpo contra este marcador têm que ser previamente fixadas e permeabilizadas, para permitir a penetração do anticorpo através da membrana celular. A fixação e permeabilização celulares são métodos que, por exporem o interior da célula, afetam a qualidade do RNA, qualidade esta que é fundamental para a realização de microarrays. Assim, uma metodologia foi desenvolvida para permitir obter RNA de elevada qualidade a partir de células fixadas, permeabilizadas, marcadas com um anticorpo fluorescente anti-KRT18 e separadas por citometria de fluxo.
Devido à dificuldade na obtenção de amostras embrionárias e fetais suficientes, a linha celular MCF-7 (que também expressa KRT18) foi usada para a o desenvolvimento e otimização deste protocolo, tendo este sido posteriormente aplicado em células da coluna vertebral fetal. O protocolo foi dividido em 3 passos:
i) fixação e permeabilização;
ii) fixação, permeabilização e marcação com anticorpo fluorescente anti-KRT18;
iii) fixação, permeabilização, marcação com anti-KRT18 e separação por citometria de fluxo.
O RNA das células MCF-7 foi extraído após cada um destes passos e a sua quantidade, grau de pureza e integridade foram analisados. Todas as experiências foram realizadas em triplicado e os reagentes usados eram certificados como livres de RNAses e DNAses.
Para reproduzir o limitado número de células existente na coluna vertebral embrionária e fetal 5x104 células MCF-7 foram usadas em cada condição experimental. Abreviadamente, as células foram cultivadas, tripsinizadas, contadas com um hemocitómetro e distribuídas pelo número adequado de tubos de micro-centrifugação. Após lavagem em PBS, as células foram incubadas com os vários agentes de fixação e permeabilização a ser testados (Quadro II) a 4ºC durante 10 minutos. Seguidamente, as células foram lavadas em PBS e incubadas a 4ºC durante 10 minutos na presença de 1.5µg/mL de anti-KRT18 conjugado diretamente com isocianato de fluoresceína (FITC) em PBS. Finalmente, as células foram ressuspendidas em PBS para separação por citometria de fluxo. IgG de rato diretamente conjugada com FITC foi usada como controlo negativo.

Separação de células da notocorda e do esclerótomo isoladas a partir da coluna vertebral fetal
Após otimização do método adequado para marcação e separação de células KRT18-positivas, células extraídas da coluna vertebral de um feto de 9 SPC foram fixadas e permeabilizadas, marcadas com FITC-anti-KRT18 e separadas por citometria de fluxo.
A citometria de fluxo foi realizada com o instrumento FACS Aria (BD Biosciences®). A excitação de partículas foi realizada com o laser azul (488nm) e a deteção com o filtro 530/30. As células separadas foram colhidas para um tubo de micro-centrifugação contendo 350µL de tampão de lise celular (RLT lysis buffer, RNeasy micro plus kit, Qiagen®).
Extração e análise da quantidade, grau de pureza e integridade do RNA e amplificação para cDNA
O RNA foi extraído com o RNeasy micro plus kit (Qiagen®). As seguintes alterações ao protocolo base foram realizadas:
i) o lisado celular foi aquecido a 37ºC antes da sua homogeneização;
ii) para eluir o RNA foi utilizada água a 60ºC;
iii) para aumentar a quantidade de RNA obtida, o eluente foi repipetado pela coluna de extração de RNA.
A quantificação e análise da pureza do RNA foram realizadas com o Nanodrop ND-1000 Spectrophotometer (Thermo Fisher Scientific®), que avalia a concentração e os rácios 260/280 e 260/230. Estes rácios avaliam o grau de pureza do RNA, que é máximo para valores próximos de 2. A integridade do RNA foi avaliada com o Algilent 2100 Bioanalyser (Algilent Technologies®), que mede o RNA integrity number (RIN), que varia entre 0 e 10 pontos, sendo 10 o valor máximo de integridade.
O RNA obtido foi amplificado para Spia® (Single Primer Isothermal Amplification) cDNA com o Ovation Pico WTA v2 kit (NuGen Technologies®), purificado com o RNeasy MinElute kit (Qiagen®) e fragmentado e marcado para análise com o Encore Biotin Module (NuGen Technologies®). Todos estes passos foram realizados de acordo com as instruções do produtor.
Validação da separação celular através da análise da reação em cadeia da polimerase em tempo real
Para determinar a precisão da separação entre células da notocorda e do esclerótomo, realizou-se a reação em cadeia da polimerase em tempo real (qRTPCR) para genes anteriormente descritos como sendo da notocorda (Quadro III). Todas as reações foram realizadas em triplicado usando o Applied Biosystem StepOnePlus® e a expressão relativa de cada gene foi normalizada à expressão do gene constitutivo glyceraldehyde-3-phosphate dehydrogenase (GAPDH), como previamente descrito[53].
Microarrays de DNA complementar (cDNA)
Para hibridização com o gene chip Human Genome U133 Plus 2.0 microarray (Affymetrix®) foram usados 2.5µg de cDNA de cada uma das amostras. O controlo de qualidade foi realizado com o software dChip[54] e a análise dos genes com expressão diferencial foi realizada com o pacote PUMA[55]. A interactive pathway analysis (Ingenuity Systems®) foi usada para identificar redes celulares, funções biológicas e reguladores dos genes com expressão diferencial entre as células da notocorda e as células do esclerótomo. Os genes com expressão diferencial foram definidos como aqueles que apresentavam um rácio logarítmico de base 2 (Log2) superior a 2 (equivalente ao quádruplo da expressão) e uma probabilidade logarítmica normalizada positiva (PPLR) <0.05 ou >0.95.
Análise estatística
A análise estatística foi realizada com o software GraphPad InStat (GraphPad Software Inc®), usando o teste de Mann-Whitney-U; valores de p<0.05 foram definidos como representativos de diferença significativa entre médias.A análise estatística foi realizada com o software GraphPad InStat (GraphPad Software Inc®), usando o teste de Mann-Whitney-U; valores de p<0.05 foram definidos como representativos de diferença significativa entre médias.
RESULTADOS
Análise morfológica da coluna vertebral embrionária e fetal
A coloração H&E identificou a presença de células da notocorda grandes e vacuoladas ocupando uma posição central na coluna embrionária e fetal. Estas células estavam organizadas continuamente ao longo do eixo do feto até à 8ª SPC, a partir da qual se encontravam restritas à região do disco intervertebral. Nesta região, e perifericamente às células da notocorda, as células do esclerótomo apresentavam uma organização lamelar característica do AF adulto. Nas regiões adjacentes, a densidade celular das células do esclerótomo percursoras do corpo vertebral era menor; no seu centro, e a partir da 10a SPC, estas tornavam-se hipertróficas, constituindo o centro de ossificação vertebral (Figura 2).
Identificação de um marcador específico para as células da notocorda
A pesquisa da literatura identificou 16 potenciais marcadores específicos das células da notocorda (Quadro IV). Destes, as moléculas BASP1, Galectin-3, CD55, CTGF, KRT8, KRT18, KRT19 e N-Cad foram selecionadas para análise imunohistoquímica da sua expressão em secções da coluna vertebral embrionária e fetal. Verificou-se que as proteínas KRT8, KRT18 e KRT19 eram específicas para as células da notocorda humana em todos os níveis vertebrais (cervical, torácico e lombar) e em todas as idades analisadas (Figura 3), tendo a KRT18 sido escolhida como o marcador a usar para separar as células da notocorda (KRT18-positivas) das células do esclerótomo (KRT18-negativas).
Desenvolvimento de uma metodologia para obtenção de RNA a partir de células marcadas com um anticorpo intracelular
i) A análise do RNA extraído a partir de células MCF-7 fixadas e permeabilizadas com diferentes agentes demonstrou uma redução significativa na sua concentração quando comparados com o controlo positivo (PBS). Os agentes Etanol/ ácido acético e 100% RNAlater foram aqueles com os quais se obteve RNA com maior grau de pureza e integridade (Figura 4), pelo que foram selecionados como os métodos de fixação e permeabilização a usar nos passos seguintes.
ii) Para avaliar o efeito da marcação intracelular com FITC-anti-KRT18, células MCF-7 foram fixadas e permeabilizadas com os dois agentes selecionados previamente e incubadas com FITCanti-KRT18 ou PBS (controlo). Verificou-se que a marcação com FITC-anti-KRT18 não exercia efeito deletério sobre a quantidade ou integridade do RNA, independentemente do agente de fixação usado previamente (etanol/ ácido acético ou RNAlater®). No entanto, o grau de pureza (analisado pelo rácio 260/230) após a fixação, permeabilização e marcação com anticorpo era significativamente inferior quando o RNAlater® era usado (Figura 5).
iii) Verificou-se que o RNA obtido após separação por citometria de fluxo de células MCF-7 fixadas e permeabilizadas com etanol/ácido acético e marcadas com FITC-anti-KRT18 apresentava concentrações e integridade significavamente superiores às obtidas quando o RNAlater® era usado como método de fixação e permeabilização (Figura 6).
Estes resultados permitiram desenvolver uma metodologia que consistia na fixação e permeabilização com etanol/ácido acético, seguida de marcação com FITC-anti-KRT18 e separação celular por citometria de fluxo e que era capaz de obter RNA com suficiente qualidade e integridade para a realização de microarrays a partir de um número limitado de células, pelo que esta foi usada para o isolamento de RNA a partir de células da coluna vertebral fetal humana.
Separação por citometria de fluxo e extração de RNA a partir de células da notocorda e do esclerótomo da coluna vertebral humana
A coluna vertebral de um feto de 9 SPC foi dissecada de todos os tecidos adjacentes e as suas células (da notocorda e do esclerótomo) foram extraídas enzimaticamente, fixadas e permeabilizadas, marcadas com FITC-anti-KRT18 e separadas por citometria de fluxo de acordo com a metodologia desenvolvida com células MCF-7. A análise citométrica do nível de fluorescência detetou 3800 eventos KRT18-positivos (células da notocorda) e 40000 eventos KRT18-negativos (células do esclerótomo) (Figura 7).
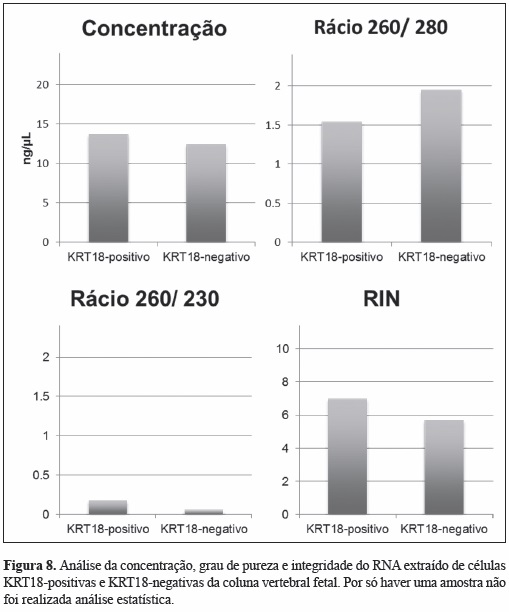
Extração de RNA a partir de células da notocorda e do esclerótomo humano
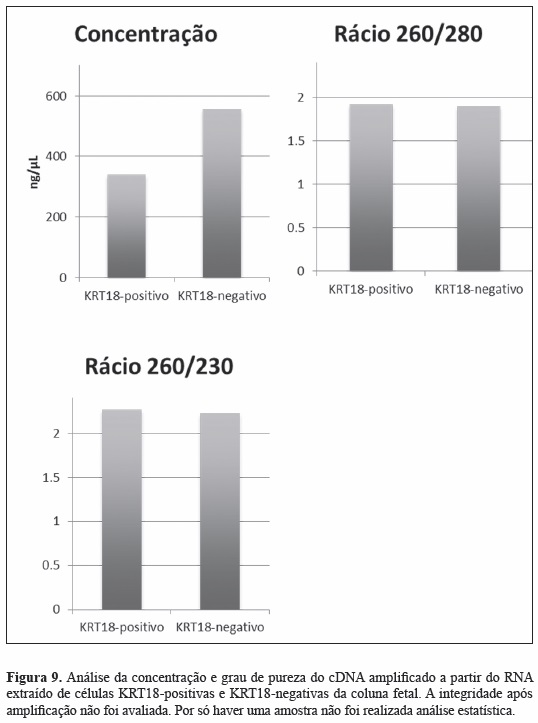
Treze microlitros de RNA com concentrações de 13.7ng/µL e 12.4ng/µL foram respetivamente obtidos a partir de células KRT18-positivas e KRT18-negativas da coluna vertebral fetal. Este RNA apresentava elevados rácio 260/280 e valores de RIN mas baixos rácio 260/230 e concentração (Figura 8). Devido à sua baixa concentração, o RNA foi amplificado para cDNA por um fator de 25 vezes nas células KRT18-positivas e 45 vezes nas células KRT18-negativas. Esta amplificação melhorou também significavamente o rácio 260/230 (Figura 9).
Validação da separação celular por reação em cadeia da polimerase em tempo real
Para validar a precisão da metodologia usada para separar células da notocorda (KRT18-positivas) das células do esclerótomo (KRT18-negativas), a expressão de genes previamente descritos como sendo da notocorda (KRT18, KRT19, T, Galectin-3 e FOXA2) foi comparada entre os dois tipos celulares. A comparação mostrou que as células KRT18-positivas têm uma expressão aumentada de todos os genes analisados, comparativamente com as células KRT18- negativas (Figura 10).
Análise dos microarrays
A análise da expressão diferencial entre as células separadas identificou 782 genes regulados positivamente e 678 genes regulados negativamente pelas células KRT18-positivas em relação às KRT18-negativas. O quadro V apresenta os 10 genes com maior expressão diferencial entre as células KRT18-positivo e KRT18-negativo (marcadores positivos) e os 10 genes com maior expressão diferencial entre as células KRT18-negativo e KRT18-positivo (marcadores negativos).
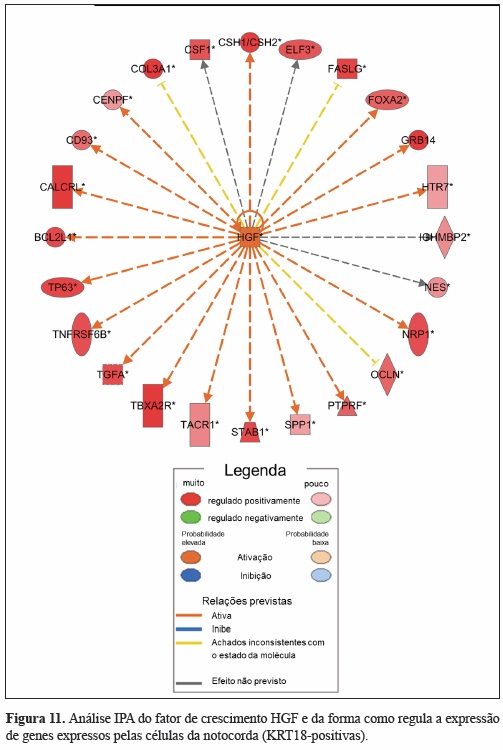
A análise IPA dos fatores a montante dos genes KRT18-positivos identificou 30 reguladores destes genes (24 previstos estar ativados e 6 inibidos) (Quadro VI). Destes, o hepatocyte growth factor (HGF) foi o fator de crescimento com probabilidade de ativação (score z) mais elevado. A análise das vias de regulação deste fator de crescimento identificou diversas moléculas com expressão diferencial aumentada nas células KRT18-positivas e envolvidas no desenvolvimento da coluna vertebral e na atividade biológica das células da notocorda (Figura 11).
DISCUSSÃO
As células da notocorda ou fatores por estas produzidos têm sido propostos como tendo um efeito anabólico e protetor contra a degeneração do disco intervertebral e o seu “desaparecimento” aquando da maturidade esquelética, tem sido sugerido como sendo o fator iniciador da degeneração discal. Deste modo, a identificação do fenótipo destas células e das moléculas que as regulam poderá elucidar a comunidade científica sobre quais os fatores que regulam a homeostasia do disco intervertebral e ajudar no desenvolvimento de novas terapêuticas biológicas e estratégias celulares para combater a sua degeneração.
Apesar de terem já sido efetuadas várias tentativas para definir o fenótipo das células da notocorda em modelos animais[56, 57, 68-72], os resultados desses estudos não são diretamente aplicáveis à investigação humana, já que o fenótipo da célula do NP, que deriva da notocorda, varia consideravelmente entre espécies[36, 57, 73]. Isto pode dever-se a diferenças de organização celular, forma e tamanho do disco intervertebral, postura e locomoção entre as diferentes espécies de mamíferos[73, 74]. A realização de estudos em humanos tem, no entanto, sido dificultada por restrições éticas e logísticas relacionadas com a aquisição de tecidos contendo células da notocorda, bem como por dificuldades técnicas relacionadas com a dimensão das colunas vertebrais embrionária e fetal e a falta de métodos adequados para separação de células para realização de estudos de expressão genética em grande escala, como microarrays. Esta lacuna tem sido uma limitação importante para o aprofundamento do conhecimento sobre o desenvolvimento, maturação e degeneração do disco intervertebral e para a descoberta de métodos para o regenerar.
Assim, os objetivos deste trabalho foram:
i) desenvolver um método para o isolamento e separação de células da notocorda (precursoras do NP) das células do esclerótomo (precursoras do AF e vértebra) que lhe são adjacentes;
ii) caraterizar o fenótipo das células da notocorda e identificar fatores envolvidos na função anabólica e protetora desempenhada por estas células no disco intervertebral.
Devido à ausência de uma demarcação clara entre os limites das células do esclerótomo e da notocorda fetais foi desenvolvida uma estratégia de separação baseada na identificação de um marcador celular específico para as células da notocorda, marcação dessas células com um anticorpo fluorescente dirigido para o marcador identificado e separação das células por citometria de fluxo.
Primeiro, a coluna vertebral fetal foi dissecada de todos os tecidos adjacentes com o objetivo de limitar o tipo de células a separar a dois tipos: células da notocorda e células do esclerótomo.
Segundo, foi realizada uma revisão extensa da literatura para identificar um marcador específico para as células da notocorda. Uma vez que há muito pouca informação sobre estas células, pretenderam identificar-se todas as moléculas que haviam sido descritas como sendo expressas por células da notocorda embrionária e fetal (em estudos de biologia do desenvolvimento), células da notocorda do NP de animais que as retêm ao longo da vida ou células do NP humano adulto (que, apesar de não possuir células com morfologia da notocorda, se pensa ser derivado desta[50]). Foram analisados todos os artigos identificados, bem como referências relevantes desses mesmos artigos e de revisões da literatura dentro da mesma área. Dezasseis potenciais marcadores foram identificados. Destes, 8 (BASP1, Galectin-3, CD55, CTGF, KRT8, KRT18, KRT19 e N-Cad) foram selecionados para validação por imunohistoquímica na coluna embrionária e fetal humana. Estas moléculas foram selecionadas por terem sido identificadas em estudos humanos e animais. Verificou-se que as KRT8, KRT18 e KRT19 eram expressas por todas as células da notocorda em todos os níveis vertebrais e em todas as idades gestacionais avaliadas.
As citoqueratinas são moléculas do citoesqueleto e a sua expressão, apesar de indicativa de um fenótipo epitelial, tem sido também observada em células derivadas da mesoderme como cardiomiócitos[75] e fibroblastos[76]. A KRT18 dimeriza com a KRT8 para formar um filamento intermediário em epitélios estratificados simples, enquanto que a KRT19 é a mais pequena citoqueratina acídica conhecida. As KRT8 e KRT18 estão envolvidas na resistência à apoptose induzida pelo TNF-a em hepatócitos[77]. De relevo para a área do disco intervertebral, estas moléculas encontram-se frequentemente presentes em tecidos submetidos a carga mecânica[78, 79], pelo que a sua presença no disco intervertebral poderá servir para ajudar a suportar a elevada pressão hidrostática existente no NP[80]. Uma vez que não se verificaram diferenças no padrão e intensidade de coloração das células da notocorda pelas três citoqueratinas, e já que nós identificámos recentemente a KRT18 como um marcador do NP adulto[36], esta foi escolhida para marcar as células da notocorda a serem separadas por citometria.
A KRT18, tal como todas as citoqueratinas é, no entanto, uma proteína intracelular, pelo que para que o anticorpo consiga penetrar através da membrana celular, é necessário fixar e permeabilizar as células. A extração de RNA de células fixadas e permeabilizadas é, contudo, complexa e as únicas tentativas para o fazer reportam à década de 90[81, 82], em que é proposto o uso de agentes de fixação e permeabilização de base alcoólica. Contudo, o RNA obtido nestes estudos não apresentava qualidade suficiente para a realização de microarrays.
Deste modo, foi desenvolvida uma metodologia para isolar RNA com elevado grau de pureza e integridade a partir de células previamente fixadas, permeabilizadas, marcadas com um anticorpo fluorescente e separadas por citometria de fluxo. Para o desenvolvimento desta metodologia foi inicialmente usada a linha celular MCF-7, tendo esta só posteriormente sido aplicada a células da coluna fetal. Para minimizar a degradação do RNA, todos os procedimentos foram realizados em ambiente livre de RNAses e o número total de passos foi reduzido, eliminando lavagens celulares que se verificaram redundantes e usando um anticorpo conjugado diretamente (e não um anticorpo primário seguido de um secundário). Inicialmente, foi avaliado o efeito sobre a qualidade do RNA de agentes de fixação e permeabilização de base alcoólica (etanol/ácido acético, 100% e 50% etanol e UM-Fix®) e não alcoólica (100% ou 50% RNAlater®), métodos adaptados a partir de estudos prévios[81-84], tendo-se verificado que dois deles (etanol/ácido acético e RNAlater®) tinham o menor impacto negativo sobre a qualidade de RNA obtida e, portanto, tendo sido escolhidos para os passos seguintes. Posteriormente, verificouse que a marcação com anticorpo fluorescente não afetava a qualidade do RNA em células fixadas e permeabilizadas com etanol/ácido acético e diminuía ligeiramente o grau de pureza do RNA em células fixadas com 100% RNAlater®. Por último, avaliouse o efeito da separação celular por citometria de fluxo, tendo-se verificado que a qualidade do RNA das células fixadas e permeabilizadas com 100% RNAlater® era drasticamente reduzida, enquanto que a das células fixadas com etanol/ácido acético não era afetada. Foi recentemente demonstrado que a radiação de campo elétrico emitida pelo citómetro de fluxo pode causar disrupção celular[85] e talvez esta seja a razão pela qual, nas células fixadas e permeabilizadas com RNAlater® o RNA é degradado. Não é clara, no entanto, a razão pela qual quando as células são fixadas com etanol/ácido acético isto não acontece mas poderá estar relacionado com a diferente natureza dos dois agentes utilizados.
Esta metodologia (fixação com etanol/ácido acético, seguida de marcação com anti-KRT18 marcado com anticorpo fluorescente e separação celular por citometria de fluxo) é uma nova estratégia para a obtenção de RNA de elevada qualidade a partir de células marcadas com um anticorpo intracelular. Esta foi a estratégia usada para identificar o fenótipo das células da notocorda humana, mas é também uma nova metodologia cuja utilidade e aplicação se estendem a qualquer área de investigação.
Utilizando o método estabelecido com a linha celular, células da coluna vertebral fetal humana foram isoladas enzimaticamente, fixadas e permeabilizadas, marcadas com KRT18 e separadas em células da notocorda (KRT18-positivas) e do esclerótomo (KRT18-negativas). Tal como previsto pela avaliação morfológica da coluna fetal, a proporção de células do esclerótomo (90.5%) foi superior à de células da notocorda (9.5%). Apesar do número limitado de células separadas, foi isolado RNA e, após amplificação para cDNA, este apresentava elevada concentração, grau de pureza e integridade. O grau de precisão da separação em células da notocorda e do esclerótomo foi confirmado pela maior expressão diferencial dos genes da notocorda KRT18, KRT19, T, Gal3, CTGF e FoxA2 pelas células KRT18-positivas do que pelas KRT18-negativas.
Os microarrays são uma técnica potente capaz de medir a expressão de todos os genes conhecidos do genoma humano numa determinada célula num tempo específico e a informação proveniente destes estudos tem sido usada para caracterizar doenças, prever a sua progressão e desenvolver novas terapêuticas[86-88]. Na área do disco intervertebral foram usados para caracterizar o fenótipo do NP adulto e para identificar novos marcadores celulares que têm sido utilizados para avaliar a correta diferenciação de células estaminais em células do NP[36, 37, 56, 57, 61, 89-92].
As células NP do adulto podem, no entanto, não ter o fenótipo adequado para regenerar o disco intervertebral já que, com a idade, estas células apresentam uma aumento da senescência[93], da morte celular mediada por autofagia[94] e da expressão de enzimas catabólicas e degradadoras da matriz extracelular[95, 96] e uma diminuição da expressão dos componentes normais da matriz extracelular[9, 97]. Por outro lado, e como detalhado anteriormente, as células da notocorda têm propriedades anabólicas e anticatabólicas, o que indica que elas, ou fatores por elas produzidos, são capazes de produzir uma matriz extracelular mais hidratada, que melhor poderá executar as funções do NP no disco intervertebral. Deste modo, a caracterização do fenótipo destas células e dos mecanismos biológicos responsáveis pela sua função são essenciais para o desenvolvimento de terapêuticas celulares para regenerar o disco intervertebral.
O estudo por microarrays realizado identificou uma lista de marcadores (positivos e negativos) com elevada expressão diferencial entre os dois tipos celulares. Apesar da validação destes marcadores numa coorte mais alargada estar ainda em curso, os dados obtidos até agora permitem uma análise detalhada de genes que poderão desempenhar papéis fundamentais na biologia das células da notocorda.
O receptor growth fator recetor-bound protein 14 (GRB14), que é uma molécula que interage com o recetor insulin growth factor 1 (IGF1), foi o marcador positivo com maior expressão diferencial entre os dois tipos celulares e poderá assumir particular relevância. O IGF1 é um fator de crescimento que aumenta a produção de proteoglicanos por células do NP bovino[98] e murídeo[99], estimula a proliferação de células do NP bovinas[100] e humanas[101] e inibe a morte celular induzida pela Il-1 em células do NP de coelhos[102]; além disso, a produção aumentada de proteoglicanos induzida pelo IGF1 em células do NP murídeo diminui com a idade[99]. É possível que a elevada produção de proteoglicanos pelas células da notocorda esteja relacionada com uma maior resposta ao IGF1 dependente do seu recetor GRB14 e que, com a maturação do disco intervertebral e o desaparecimento da células da notocorda (e do recetor GRB14 por elas expresso) esta capacidade seja perdida. Esta hipótese poderá ser testada pela avaliação da expressão de GRB14 no NP humano de diferentes idades.
Por outro lado, as moléculas FAS e CD40 (recetores do citocina pró-inflamatória TNF) foram identificadas como marcadores negativos das células da notocorda. A baixa expressão destes recetores nas células da notocorda indica que estas células poderão não responder a esta citocina, podendo estar protegidas dos efeitos pró-inflamatórios e catabólicos do TNF-a verificados durante a degeneração discal[102, 103].
Além de fornecer uma extensa lista de genes e marcadores celulares, a análise de microarrays por IPA permite a identificação de vias e mecanismos celulares, bem como de reguladores a montante das células em estudo. O IPA combina as listas de genes identificados no estudo com bases de dados da literatura científica, prevendo relações entre moléculas, mecanismos, e fatores reguladores da função das células em investigação. A análise dos fatores a montante da lista de marcadores positivos das células da notocorda identificou o HGF como sendo o fator de crescimento com maior probabilidade de ativar os seus genes, e portanto, algumas das funções destas células. O HGF é um regulador positivo do GRB14, do FoxA2 e do neuropilin 1 (NRP1). O FoxA2 é um gene que, em conjunto com o FoxA1 é crucial para o desenvolvimento do NP[63]. O NRP-1 é um recetor do semaphorin 3A (Sema3A9) que está envolvido da repulsão de neurónios da notocorda[104] e que atua como barreira para o crescimento de nervos para o disco intervertebral saudável[105]. Estes dados sugerem que o fator de crescimento HGF tem um papel importante na regulação das células da notocorda, regulando positivamente a expressão de genes importantes para o desenvolvimento e função adequada do NP.
CONCLUSÃO
Este é o primeiro estudo a identificar e separar células da notocorda das células do esclerótomo na coluna vertebral humana e a caracterizar o fenótipo, mecanismos e vias de regulação dessas células. Para o fazer, foi desenvolvida uma metodologia inovadora que permite extrair RNA de elevada qualidade a partir de um número limitado de células fixadas, permeabilizadas, marcadas com um anticorpo intracelular e separadas por citometria de fluxo. Esta metodologia tem aplicações que vão para além da área do disco intervertebral.
Os genes identificados representam um conjunto de marcadores positivos e negativos das células da notocorda e constituem a assinatura genética destas células, que poderá agora ser usada para estudar o desenvolvimento destas células e o seu destino com a maturação do disco intervertebral humano, mas também para avaliar a diferenciação de células estaminais em células da notocorda.
O fator de crescimento HGF foi identificado como um regulador a montante das células da notocorda e poderá, sozinho ou em combinação com outros fatores de crescimento, ser responsável por algumas das funções atribuídas a estas células.
REFERÊNCIAS BIBLIOGRÁFICAS
1. Hoy D. A systematic review of the global prevalence of low back pain. Arthritis Rheum. 2012; 64 (6): 2028-2037 [ Links ]
2. Balague F. Non-specific low back pain. Lancet. 2012; 379 (9814): 482-491 [ Links ]
3. Cheung KM. Prevalence and pattern of lumbar magnetic resonance imaging changes in a population study of one thousand forty-three individuals. Spine (Phila Pa 1976). 2009; 34 (9): 934-940 [ Links ]
4. Livshits G. Lumbar disc degeneration and genetic factors are the main risk factors for low back pain in women: the UK Twin Spine Study. Ann Rheum Dis. 2011; 70 (10): 1740-1745 [ Links ]
5. Bogduk N. The lumbar disc and low back pain. Neurosurg Clin N Am. 1991; 2 (4): 791-806 [ Links ]
6. Freemont AJ. Nerve ingrowth into diseased intervertebral disc in chronic back pain. Lancet. 1997; 350 (9072): 178-181 [ Links ]
7. Humzah MD, Soames RW. Human intervertebral disc: structure and function. Anat Rec. 1988; 220 (4): 337-356 [ Links ]
8. Anderson DG. Cellular therapy for disc degeneration. Spine (Phila Pa 1976). 2005; 30 (17): 14-19 [ Links ]
9. Sive JI. Expression of chondrocyte markers by cells of normal and degenerate intervertebral discs. Mol Pathol. 2002; 55 (2): 91-97 [ Links ]
10. Videman T, Nurminen M. The occurrence of anular tears and their relation to lifetime back pain history: a cadaveric study using barium sulfate discography. Spine (Phila Pa 1976). 2004; 29 (23): 2668-2676 [ Links ]
11. Kirkaldy-Willis WH. Pathology and pathogenesis of lumbar spondylosis and stenosis. Spine (Phila Pa 1976). 1978; 3 (4): 319-328 [ Links ]
12. Williams FM. Progression of lumbar disc degeneration over a decade: a heritability study. Ann Rheum Dis. 2011; 70 (7): 1203-1207 [ Links ]
13. Errico TJ. Lumbar disc arthroplasty. Clin Orthop Relat Res. 2005; 435: 106-117 [ Links ]
14. Lund T, Oxland TR. Adjacent Level Disk Disease-Is it Really a Fusion Disease?. Orthop Clin North Am. 2011; 42 (4): 529-541 [ Links ]
15. Ahmad Z. Exploring the application of stem cells in tendon repair and regeneration. Arthroscopy. 2012; 28 (7): 1018-1029 [ Links ]
16. Pereira H. Tissue engineering and regenerative medicine strategies in meniscus lesions. Arthroscopy. 2011; 27 (12): 1706-1719 [ Links ]
17. Matsumoto T. Acceleration of tendon-bone healing of anterior cruciate ligament graft using autologous ruptured tissue. Am J Sports Med. 2012; 40 (6): 1296-1302 [ Links ]
18. Veronesi F. Clinical use of bone marrow, bone marrow concentrate, and expanded bone marrow mesenchymal stem cells in cartilage disease. Stem Cells Dev. 2013; 22 (2): 181-192 [ Links ]
19. Griffin M, Iqbal SA, Bayat A. Exploring the application of mesenchymal stem cells in bone repair and regeneration. J Bone Joint Surg Br. 2011; 93 (4): 427-434 [ Links ]
20. Chen FH, Rousche KT, Tuan RS. Technology Insight: adult stem cells in cartilage regeneration and tissue engineering. Nat Clin Pract Rheumatol. 2006; 2 (7): 373-382 [ Links ]
21. Barry FP, Murphy JM. Mesenchymal stem cells: clinical applications and biological characterization. Int J Biochem Cell Biol. 2004; 36 (4): 568-584 [ Links ]
22. Pittenger MF. Multilineage potential of adult human mesenchymal stem cells. Science. 1999; 284 (5411): 143-147 [ Links ]
23. Sonoyama W. Mesenchymal stem cell-mediated functional tooth regeneration in swine. PLoS One. 2006; 1: 79 [ Links ]
24. Caplan AI. Adult mesenchymal stem cells for tissue engineering versus regenerative medicine. J Cell Physiol. 2007; 213 (2): 341-347 [ Links ]
25. Noel D, Djouad F, Jorgense C. Regenerative medicine through mesenchymal stem cells for bone and cartilage repair. Curr Opin Investig Drugs. 2002; 3 (7): 1000-1004 [ Links ]
26. Arinzeh TL. Mesenchymal stem cells for bone repair: preclinical studies and potential orthopedic applications. Foot Ankle Clin. 2005; 10 (4): 651-665 [ Links ]
27. Hong L, Colpan A, Peptan IA. Modulations of 17-beta estradiol on osteogenic and adipogenic differentiations of human mesenchymal stem cells. Tissue Eng. 2006; 12 (10): 2747-2753 [ Links ]
28. Helder MN. Stem cells from adipose tissue allow challenging new concepts for regenerative medicine. Tissue Eng. 2007; 13 (8): 1799-808 [ Links ]
29. Trubiani O. Morphological and cytofluorimetric analysis of adult mesenchymal stem cells expanded ex vivo from periodontal ligament. Int J Immunopathol Pharmacol. 2005; 18 (2): 213-221 [ Links ]
30. Henriksson HB. Transplantation of human mesenchymal stems cells into intervertebral discs in a xenogeneic porcine model. Spine (Phila Pa 1976). 2009; 34 (2): 141-148 [ Links ]
31. Richardson SM. The differentiation of bone marrow mesenchymal stem cells into chondrocyte-like cells on poly- L-lactic acid (PLLA) scaffolds. Biomaterials. 2006; 27 (22): 4096-4078 [ Links ]
32. Richardson SM. Human mesenchymal stem cell differentiation to NP-like cells in chitosan-glycerophosphate hydrogels. Biomaterials. 2008; 29 (1): 85-93 [ Links ]
33. Risbud MV. Differentiation of mesenchymal stem cells towards a nucleus pulposus-like phenotype In vitro: implications for cell-based transplantation therapy. Spine (Phila Pa 1976). 2004; 29 (23): 2627-2632 [ Links ]
34. Stoyanov JV. Role of hypoxia and growth and differentiation factor-5 on differentiation of human mesenchymal stem cells towards intervertebral nucleus pulposus-like cells. Eur Cell Mater. 2011; 21: 533-547 [ Links ]
35. Strassburg S. Bi-directional exchange of membrane components occurs during co-culture of mesenchymal stem cells and nucleus pulposus cells. PLoS One. 2012; 7 (3) [ Links ]
36. Minogue BM. Characterization of the human nucleus pulposus cell phenotype and evaluation of novel marker gene expression to define adult stem cell differentiation. Arthritis Rheum. 2010; 62 (12): 3695-3705 [ Links ]
37. Powers KA. Identification of cell surface-specific markers to target human nucleus pulposus cells: expression of carbonic anhydrase XII varies with age and degeneration. Arthritis Rheum. 2011; 63 (12): 3876-3886 [ Links ]
38. Walmsley R. The development and growth of the intervertebral disc. Edinb Med J. 1953; 60 (8): 341-364 [ Links ]
39. Aguiar DJ, Johnson SL, Oegema TR. Notochordal cells interact with nucleus pulposus cells: regulation of proteoglycan synthesis. Exp Cell Res. 1999; 241 (1): 129-137 [ Links ]
40. Hunter CJ, Matyas JR, Duncan NA. The threedimensional architecture of the notochordal nucleus pulposus: novel observations on cell structures in the canine intervertebral disc. J Anat. 2003; 202 (3): 279-291 [ Links ]
41. Hunter CJ, Matyas JR, Duncan NA. The functional significance of cell clusters in the notochordal nucleus pulposus: survival and signaling in the canine intervertebral disc. Spine (Phila Pa 1976). 2004; 29 (10): 1099-1104 [ Links ]
42. Hunter CJ, Matyas JR, Duncan NA. Cytomorphology of notochordal and chondrocytic cells from the nucleus pulposus: a species comparison. J Anat. 2004; 205 (5): 357-362 [ Links ]
43. Cappello R. Notochordal cell produce and assemble extracellular matrix in a distinct manner, which may be responsible for the maintenance of healthy nucleus pulposus. Spine (Phila Pa 1976). 2006; 31 (8): 873-883 [ Links ]
44. Erwin WM. Nucleus pulposus notochord cells secrete connective tissue growth factor and up-regulate proteoglycan expression by intervertebral disc chondrocytes. Arthritis Rheum. 2006; 54 (12): 3859-3867 [ Links ]
45. Erwin WM. Notochordal cells protect nucleus pulposus cells from degradation and apoptosis: implications for the mechanisms of intervertebral disc degeneration. Arthritis Res Ther. 2011; 13 (6): 215 [ Links ]
46. Hay ED. The mesenchymal cell, its role in the embryo, and the remarkable signaling mechanisms that create it. Dev Dyn. 2005; 233 (3): 706-720 [ Links ]
47. Aszodi A. Collagen II is essential for the removal of the notochord and the formation of intervertebral discs. J Cell Biol. 1998; 143 (5): 1399-1412 [ Links ]
48. Peacock A. Observations on the prenatal development of the intervertebral disc in man. J Anat. 1951; 85 (3): 260-274 [ Links ]
49. Piper K. Beta cell differentiation during early human pancreas development. J Endocrinol. 2004; 181 (1): 11-23 [ Links ]
50. Choi KS, Cohn MJ, Harfe BD. Harfe, Identification of nucleus pulposus precursor cells and notochordal remnants in the mouse: implications for disk degeneration and chordoma formation. Dev Dyn. 2008; 237 (12): 3953-3958 [ Links ]
51. Guehring T. Notochordal intervertebral disc cells: sensitivity to nutrient deprivation. Arthritis Rheum. 2009; 60 (4): 1026-1034 [ Links ]
52. Yamada H. Messenger RNA quantification after fluorescence-activated cell sorting using in situ hybridization. Cytometry A. 2010; 77 (11): 1032-1037 [ Links ]
53. Schmittgen TD, Livak KJ. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 2008; 3 (6): 1101-1108 [ Links ]
54. Li C, Wong WH. Model-based analysis of oligonucleotide arrays: expression index computation and outlier detection. Proc Natl Acad Sci U S A. 2001; 98 (1): 31-36 [ Links ]
55. Pearson RD. puma: a Bioconductor package for propagating uncertainty in microarray analysis. BMC Bioinformatics. 2009; 10: 211 [ Links ]
56. Tang X, Jing L, Chen J. Changes in the molecular phenotype of nucleus pulposus cells with intervertebral disc aging. PLoS One. 2012; 7 (12) [ Links ]
57. Minogue BM. Transcriptional profiling of bovine intervertebral disc cells: implications for identification of normal and degenerate human intervertebral disc cell phenotypes. Arthritis Res Ther. 2010; 12 (1): 22 [ Links ]
58. Chen J, Richardson WJ, Brown L, Setton LA. Gene expression profiling reveals age and zonal-specific differences in intervertebral disc tissues during ageing. Trans Orthop Res Soc. 2007; [ Links ]
59. Chen J. Expression of laminin isoforms, receptors, and binding proteins unique to nucleus pulposus cells of immature intervertebral disc. Connect Tissue Res. 2009; 50 (5): 294-306 [ Links ]
60. Gilchrist CL. Nucleus pulposus cell-matrix interactions with laminins. Eur Cell Mater. 2011; 21: 523-532 [ Links ]
61. Tang X. Identifying the molecular phenotype of cells in the human intervertebral disc reveals the existence of a unique notochordal-like cell population. Orthopaedic Research Society 2013 Annual Meeting; San Antonio, Texas; 2013. [ Links ]
62. Leung V. Defining the phenotype and molecular signatures of notochordal nucleus pulposus cells. World Forum for Spine Research (WFSR 2010); Montreal, Canada: The Intervertebral Disc; 2010.
63. Maier JA, Lo Y, Harfe BD. Foxa1 and Foxa2 are required for formation of the intervertebral discs. PLoS One. 2013; 8 (1) [ Links ]
64. Gotz W. Detection and distribution of the carbohydrate binding protein galectin-3 in human notochord, intervertebral disc and chordoma. Differentiation. 1997; 62 (3): 149-157 [ Links ]
65. Weiler C. Immunohistochemical identification of notochordal markers in cells in the aging human lumbar intervertebral disc. Eur Spine J. 2010; 19 (10): 1761-1770 [ Links ]
66. Gotz W. Intermediate filament typing of the human embryonic and fetal notochord. Cell Tissue Res. 1995; 280 (2): 455-462 [ Links ]
67. McCann MR. Tracing notochord-derived cells using a Noto-cre mouse: implications for intervertebral disc development. Dis Model Mech. 2012; 5 (1): 73-82 [ Links ]
68. Chen J, Yan W, Setton LA. Molecular phenotypes of notochordal cells purified from immature nucleus pulposus. Eur Spine J. 2006; 15 (3): 303-311 [ Links ]
69. Kim JH. Differentiation of intervertebral notochordal cells through live automated cell imaging system In vitro. Spine (Phila Pa 1976). 2009; 24 (23): 2486-2493 [ Links ]
70. Oguz E. Galectin-3 expression in the intervertebral disc: a useful marker of the notochord phenotype?. Spine (Phila Pa 1976). 2007; 32 (1): 9-16 [ Links ]
71. Poiraudeau S. Phenotypic characteristics of rabbit intervertebral disc cells. Comparison with cartilage cells from the same animals. Spine (Phila Pa 1976). 1999; 24 (9): 837-844 [ Links ]
72. Rastogi A. Environmental regulation of notochordal gene expression in nucleus pulposus cells. J Cell Physiol. 2009; 220 (3): 698-705 [ Links ]
73. Rodrigues-Pinto R, Richardson SM, Hoyland JA. Identification of novel nucleus pulposus markers: Interspecies variations and implications for cell-based therapiesfor intervertebral disc degeneration. Bone Joint Res. 2013; 2 (8): 169-178 [ Links ]
74. Alini M. Are animal models useful for studying human disc disorders/degeneration?. Eur Spine J. 2008; 17 (1): 2-19 [ Links ]
75. Kuruc N, Franke WW. Transient coexpression of desmin and cytokeratins 8 and 18 in developing myocardial cells of some vertebrate species. Differentiation. 1988; 38 (3): 177-193 [ Links ]
76. von Koskull H, Virtanen I. Induction of cytokeratin expression in human mesenchymal cells. J Cell Physiol. 1987; 133 (2): 321-329 [ Links ]
77. Caulin C. Keratin-dependent, epithelial resistance to tumor necrosis factor-induced apoptosis. J Cell Biol. 2000; 149 (1): 17-22 [ Links ]
78. Bordeleau F. Keratin contribution to cellular mechanical stress response at focal adhesions as assayed by laser tweezers. Biochem Cell Biol. 2008; 86 (4): 352-359 [ Links ]
79. Flitney EW. Insights into the mechanical properties of epithelial cells: the effects of shear stress on the assembly and remodeling of keratin intermediate filaments. FASEB J. 2009; 23 (7): 2110-2119 [ Links ]
80. Hunter CJ, Matyas JR, Duncan NA. The notochordal cell in the nucleus pulposus: a review in the context of tissue engineering. Tissue Eng. 2003; 9 (4): 667-677 [ Links ]
81. Diez C, Bertsch G, Simm A. Isolation of full-size mRNA from cells sorted by flow cytometry. J Biochem Biophys Methods. 1999; 40 (3): 69-80 [ Links ]
82. Esser C. Isolation of full-size mRNA from ethanolfixed cells after cellular immunofluorescence staining and fluorescence-activated cell sorting (FACS). Cytometry. 1995; 21 (4): 382-386 [ Links ]
83. Barrett MT. High-quality RNA and DNA from flow cytometrically sorted human epithelial cells and tissues. Biotechniques. 2002; 32 (4): 888-896 [ Links ]
84. Lamoreaux L, Roederer M, Koup R. Intracellular cytokine optimization and standard operating procedure. Nat Protoc. 2006; 1 (3): 1507-1516 [ Links ]
85. Li Q. Comparison of the sorting efficiency and influence on cell function between the sterile flow cytometry and immunomagnetic bead purification methods. Prep Biochem Biotechnol. 2013; 43 (2): 197-206 [ Links ]
86. Karlsson C. Genome-wide expression profiling reveals new candidate genes associated with osteoarthritis. Osteoarthritis Cartilage. 2010; 18 (4): 581-592 [ Links ]
87. Saito T. Transcriptional regulation of endochondral ossification by HIF-2alpha during skeletal growth and osteoarthritis development. Nat Med. 2010; 16 (6): 678-686 [ Links ]
88. Yang S. Hypoxia-inducible factor-2alpha is a catabolic regulator of osteoarthritic cartilage destruction. Nat Med. 2010; 16 (6): 687-693 [ Links ]
89. Fujita N. CD24 is expressed specifically in the nucleus pulposus of intervertebral discs. Biochem Biophys Res Commun. 2005; 338 (4): 1890-1896 [ Links ]
90. Lee CR. A phenotypic comparison of intervertebral disc and articular cartilage cells in the rat. Eur Spine J. 2007; 16 (12): 2174-2185 [ Links ]
91. Sakai D. Differential phenotype of intervertebral disc cells: microarray and immunohistochemical analysis of canine nucleus pulposus and anulus fibrosus. Spine (Phila Pa 1976). 2009; 34 (14): 1448-1556 [ Links ]
92. Smolders LA. Gene expression profiling of early intervertebral disc degeneration reveals a down-regulation of canonical Wnt signaling and caveolin-1 expression: implications for development of regenerative strategies. Arthritis Res Ther. 2013; 15 (1): 23 [ Links ]
93. Maitre CL Le, Freemont AJ, Hoyland JA. Accelerated cellular senescence in degenerate intervertebral discs: a possible role in the pathogenesis of intervertebral disc degeneration. Arthritis Res Ther. 2007; 9 (3): 45 [ Links ]
94. Ye W. Age-related increases of macroautophagy and chaperone-mediated autophagy in rat nucleus pulposus. Connect Tissue Res. 2011; 52 (6): 472-478 [ Links ]
95. Maitre CL Le, Freemont AJ, Hoyland JA. Localization of degradative enzymes and their inhibitors in the degenerate human intervertebral disc. J Pathol. 2004; 204 (1): 47-54 [ Links ]
96. Maitre CL Le, Freemont AJ, Hoyland JA. The role of interleukin-1 in the pathogenesis of human intervertebral disc degeneration. Arthritis Res Ther. 2005; 7 (4): 732-745 [ Links ]
97. Pearce RH, Grimmer BJ, Adams ME. Grimmer, and M.E. Adams, Degeneration and the chemical composition of the human lumbar intervertebral disc. J Orthop Res. 1987; 5 (2): 198-205 [ Links ]
98. Osada R. Autocrine/paracrine mechanism of insulinlike growth factor-1 secretion, and the effect of insulinlike growth factor-1 on proteoglycan synthesis in bovine intervertebral discs. J Orthop Res. 1996; 14 (5): 690-699 [ Links ]
99. Okuda S. Mechanisms of age-related decline in insulinlike growth factor-I dependent proteoglycan synthesis in rat intervertebral disc cells. Spine (Phila Pa 1976). 2001; 26 (22): 2421-2426 [ Links ]
100. Pratsinis H, Kletsas D. PDGF, bFGF and IGF-I stimulate the proliferation of intervertebral disc cells In vitro via the activation of the ERK and Akt signaling pathways. Eur Spine J. 2007; 16 (11): 1858-1866 [ Links ]
101. Zhang R, Ruan D, Zhang C. Effects of TGF-beta1 and IGF-1 on proliferation of human nucleus pulposus cells in medium with different serum concentrations. J Orthop Surg Res. 2006; 1: 9 [ Links ]
102. Zhang CC. Effects of IGF-1 on IL-1beta-induced apoptosis in rabbit nucleus pulposus cells In vitro. Mol Med Rep. 2013; 7 (2): 441-444 [ Links ]
103. Olmarker K, Larsson K. Tumor necrosis factor alpha and nucleus-pulposus-induced nerve root injury. Spine (Phila Pa 1976). 1998; 23 (23): 2538-2544 [ Links ]
104. Masuda T. Developmental regulation of notochordderived repulsion for dorsal root ganglion axons. Mol Cell Neurosci. 2004; 25 (2): 217-227 [ Links ]
105. Tolofari SK. Expression of semaphorin 3A and its receptors in the human intervertebral disc: potential role in regulating neural ingrowth in the degenerate intervertebral disc. Arthritis Res Ther. 2010; 12 (1): 1 [ Links ]
Conflito de interesse:
Nada a declarar
Serviço de Ortopedia
Centro Hospitalar do Porto
Hospital de Santo António
Largo Prof. Abel Salazar
4099-001 Porto
Portugal
ric_pinto@hotmail.com
Data de Submissão: 2013-09-01
Data de Aceitação: 2013-11-01